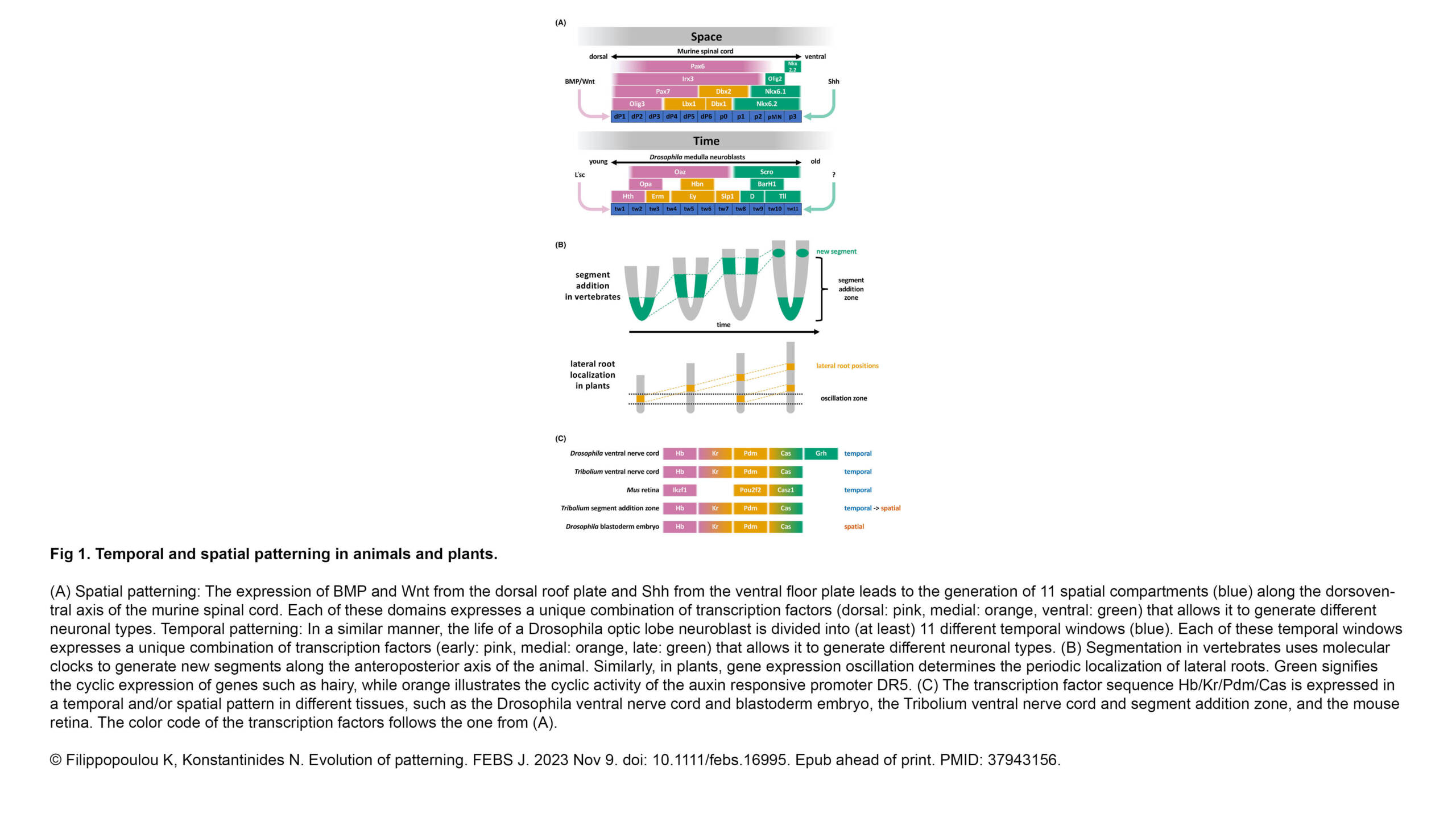
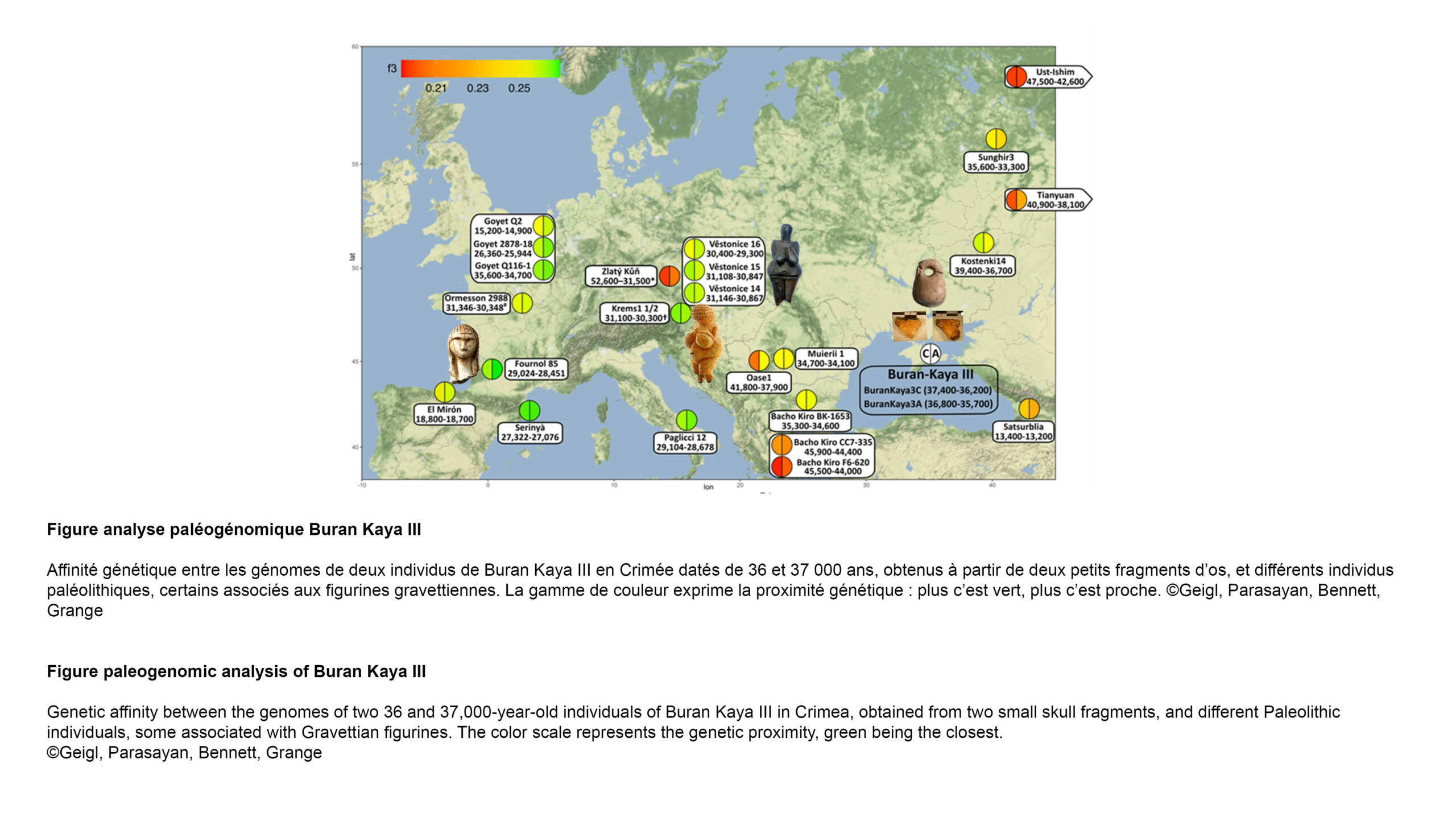
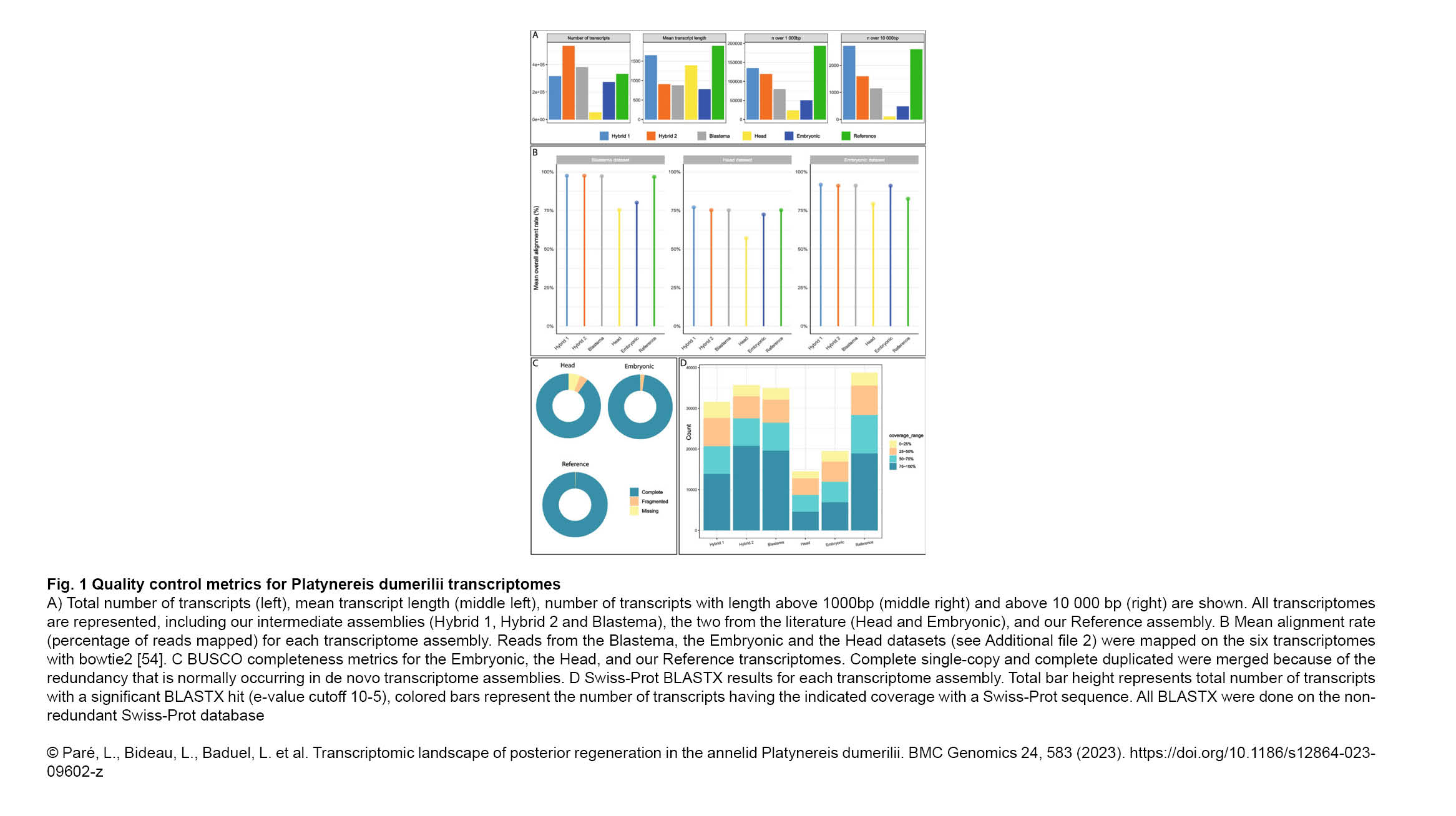
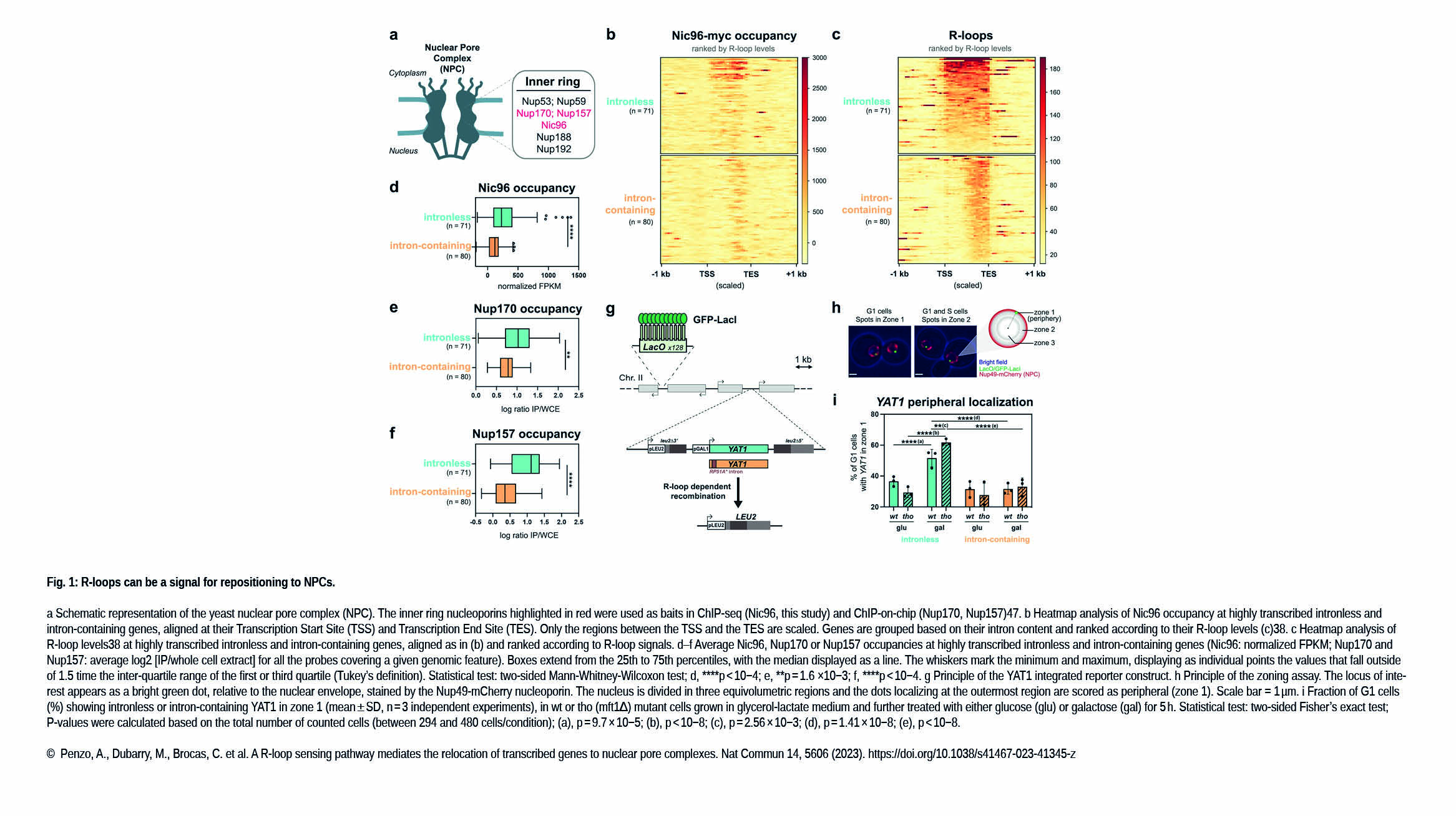
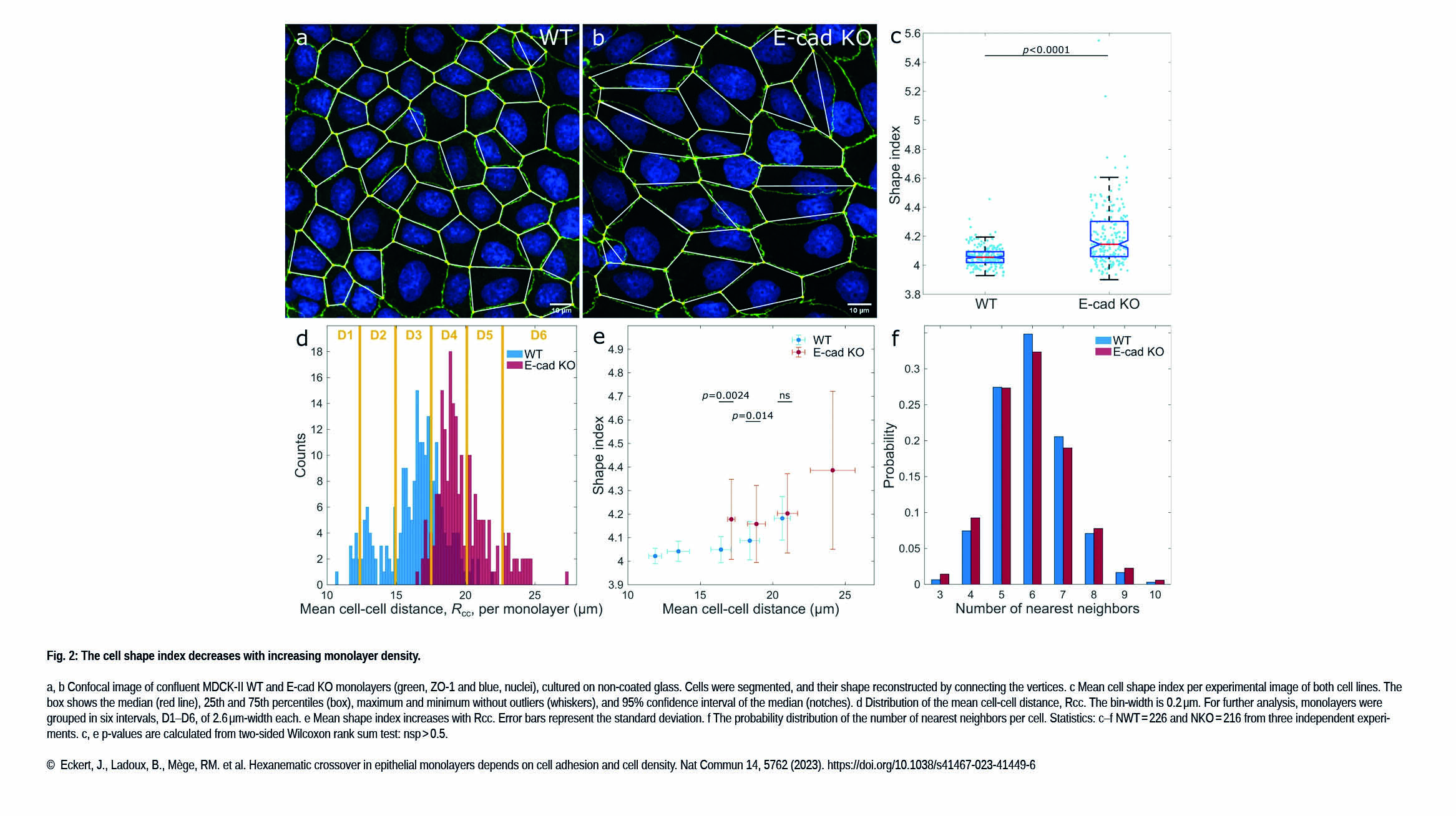
L'équipe Konstantinides a publié un nouvel article dans The FEBS Journal :
Evolution of patterning
Résumé :
Developing tissues are patterned in space and time; this enables them to differentiate their cell types and form complex structures to support different body plans. Although space and time are two independent entities, there are many examples of spatial patterns…