enSCORE : engineered spinal and cortical organoid core
 Plateforme de l’Université Paris Cité (financée par le Labex « Who Am I? »).
Plateforme de l’Université Paris Cité (financée par le Labex « Who Am I? »).
enSCORE propose un support technique et scientifique aux chercheurs pour la génération de modèles organoïdes neuraux (spinaux et corticaux) dérivés de cellules pluripotentes induites.
Mots-clés : organoïdes, iPSC, pathologie, neurobiologie du développement


+33 (0)1 57 27 80 62 contact 342b L2 Cell Culture : 315ba
La plateforme enSCORE a été créée en 2021 pour apporter un support à un consortium interdisciplinaire pour adresser des questions complexes de neurobiologie du développement. En effet, les modèles d’organoïdes dérivés de cellules souches pluripotentes induites (iPSCs) sont à ce jour les seuls modèles de développement prénatal du cerveau humain permettant de combiner des approches génétiques et pharmacologiques.
La plateforme est financée par le Labex Université Paris Cité « Who am I ? » dans le cadre du projet transversal (Organoïdes neuraux pour étudier les interactions entre les signaux mécaniques et transcriptionnels qui sous-tendent le neurodéveloppement normal et pathologique)
Les cellules pluripotentes induites humaines utilisées dérivent de cellules de donneurs sains ou de patients présentant des pathologies neurodéveloppementales. Elles sont commerciales (lignée WTC11 du Coriell Institute) ou générées dans des plateformes dédiées (Institut Imagine, Plateforme iPSC Nantes).
Les missions de la plateforme enSCORE sont :
– L’optimisation, la standardisation et la caractérisation de cultures d’iPSC humaines.
La plateforme a mis en place et offre un soutien et une formation à la culture de cellules iPS humaines ainsi qu’une caractérisation de la qualité de ces cellules (absence de mycoplasmes, marqueurs de pluripotence, intégrité chromosomique)
– Le design et la génération de lignées transgéniques par édition du génome.
En interaction avec les membres des équipes intéressées, la plateforme met en place des procédures de modélisations de pathologies, en reproduisant les mutations présentes chez les patients par des approches d’édition du génome avec les systèmes CRISPR/Cas dans des lignées d’iPSCs.
– La production et la caractérisation d’organoïdes neuraux (cérébraux et spinaux)
La plateforme emploie des protocoles variés (commerciaux, publiés, ou mis au point par des collaborateurs) pour générer les organoïdes cérébraux et spinaux et forme les utilisateurs.
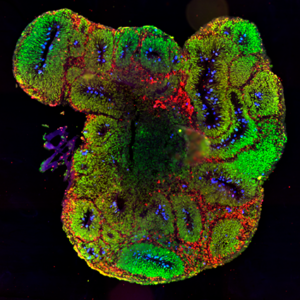
Les organoïdes générés sont phénotypés par analyses de coupes fines au cryostat et marquage par immunofluorescence ainsi que par cytométrie en flux.
La plateforme a obtenu une agrémentation à travailler sur les cellules iPSC humaines sous les CODECOH N° DC-2021-4363 et DC-2021-4446.
Membres
 Sandrine ADIBA, Responsable plateforme enSCORE, PFT/enSCORE+33 (0)1 57 27 80 62, bureau 342B
Sandrine ADIBA, Responsable plateforme enSCORE, PFT/enSCORE+33 (0)1 57 27 80 62, bureau 342B Mikaelle BOCEL, Ingénieure de plateforme, PFT/enSCORE+33 (0)1 57 27 80 62, bureau 342B
Mikaelle BOCEL, Ingénieure de plateforme, PFT/enSCORE+33 (0)1 57 27 80 62, bureau 342B Kamal BOUHALI, Responsable plateforme enSCORE, PFT/enSCORE+33 (0)1 57 27 80 62, bureau 342B
Kamal BOUHALI, Responsable plateforme enSCORE, PFT/enSCORE+33 (0)1 57 27 80 62, bureau 342B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
Responsables scientifiques:
Valerie Doye – Directrice de Recherche, Institut Jacques Monod
Valerie Mezger – Directrice de Recherche, Unité Epigénétique et Destin Cellulaire
Vanessa Ribes – Chargée de Recherche, Institut Jacques Monod
Stagiaires accueillis
Monia Abbas – Stagiaire BTS1 (16/05/22-01/07/22)
La plateforme dispose d’une pièce de culture cellulaire de confinement L2 complètement équipée pour la culture des cellules iPS et la différentiation des organoïdes.
– deux PSM réservables en ligne (https://ijm.requea.com/page/PlannigAUT)
– deux incubateurs CO2
Les équipements spécifiques comprennent notamment
– une loupe binoculaire (Dynascope Lynx, Vision Engineering) placée sous PSM permettant l’observation et la manipulation stérile des cultures. Cette loupe est équipée d’un système vidéo relié à un écran permettant la prise de photos/vidéos et permet la formation aisée des utilisateurs de la plateforme.
– Un agitateur orbital (Dutscher) nécessaire à la différentiation des organoïdes cérébraux est également installé dans l’un des incubateurs.
Les membres de la plateforme et ses utilisateurs bénéficient également de l’ensemble des équipements de l’Institut Jacques Monod notamment :
– un cryostat Leica CM3050S (datant de 2004)
Après formation par Kamal Bouhali, Mikaelle Bocel ou Vanessa Ribes ce système est réservable en ligne (https://ijm.requea.com/page/PlanningMIC#)
– un système de transfection (Nucleofector 4D) permettant la nucléofection des lignées iPSC et la génération des lignées transgéniques.
Après formation par Kamal Bouhali, ce système est réservable en ligne (https://ijm.requea.com/page/PlannigAUT)
Dans lesquelles la plateforme est remerciée :
– CBP-HSF2 structural and functional interplay in Rubinstein-Taybi neurodevelopmental disorder
Aurélie de THONEL, Johanna K. AHLSKOG, Kevin DAUPIN, Véronique DUBREUIL, Jérémy BERTHELET, Carole CHAPUT, Geoffrey PIRES, Camille LEONETTI, Ryma ABANE, Lluís CORDÓN BARRIS, Isabelle LERAY, Anna L. AALTO, Sarah NACERI, Marine CORDONNIER, Carène BENASOLO, Matthieu SANIAL, Agathe DUCHATEAU, Anniina VIHERVAARA, Mikael C. PUUSTINEN, Federico MIOZZO, Patricia FERGELOT, Élise LEBIGOT, Alain VERLOES, Pierre GRESSENS, Didier LACOMBE, Jessica GOBBO, Carmen GARRIDO, Sandy D. WESTERHEIDE Laurent DAVID, Michel PETITJEAN, Olivier TABOUREAU, Fernando RODRIGUES-LIMA, Sandrine PASSEMARD, Délara SABÉRAN-DJONEIDI, Laurent NGUYEN, Madeline LANCASTER, Lea SISTONEN, Valérie MEZGER
Projet transversal du Labex “Who am I?” Organoïdes neuraux pour étudier les interactions entre les signaux mécaniques et transcriptionnels qui sous-tendent le neurodéveloppement normal et pathologique
Autres collaborations
Equipe de Corinne Antignac (Institut IMAGINE, Paris)
Plateforme iPS, Nathalie Lefort (Institut IMAGINE, Paris)
Stéphane Nedelec (Institut du Fer à Moulin, Paris)
Didier Lacombe et Julien van Gils (CHU Bordeaux)
Sandrine Passemard et Alain Verloes, (Hôpital Robert Debré, Paris)
Madeline LANCASTER (LMB, Cambridge, UK)
Laurent Nguyen (Université de Liège, Belgique)
Auguste Genovesio (IBENS, Paris)

Le colloque sur les organoïdes cérébraux et spinaux est organisé par la plateforme enSCORE de l’Université Paris Cité (Labex « Who am I ? », Institut Jacques Monod, Epigénétique et Destin Cellulaire) et le DIM C-BRAINS.
Elle se concentre sur des modèles 3D innovants du développement du système nerveux dans des organoïdes dérivés de cellules souches pluripotentes humaines. Au cours d’une journée, l’objectif est de mettre en lumière les avancées dans trois domaines de recherche basés sur ces modèles : la modélisation des pathologies du développement neurologique, la génération de modèles multi-tissus (contenant plusieurs tissus en interaction) et l’étude des mécanismes moléculaires et cellulaires du développement humain.
Nous avons invité deux chercheurs de renommée mondiale à présenter leurs travaux sur les organoïdes cérébraux en tant qu’orateurs principaux : Orly Reiner (Institut Weizmann, Israël) et Alice Davy (CBI, Toulouse).
Cliquez ici pour le programme détaillé.

