Plateforme ProteoSeine de l’Institut Jacques Monod
ProteoSeine est la plateforme protéomique de l’Institut Jacques Monod, UMR 7592 Université de Paris Cité – CNRS. Elle est localisée depuis début 2009 dans le bâtiment Buffon au 15 rue Hélène Brion, 75205 Paris CEDEX 13 – France. Les activités de la plateforme incluent du service analytique, de la recherche et du développement technique et méthodologique ainsi qu’une veille technologique active. Dans ce contexte, elle utilise les approches les plus modernes de la spectrométrie de masse pour l’étude des constituants protéiques des systèmes vivants.
Sa mission est d’offrir à la communauté des chercheurs en biologie des compétences et services à l’état de l’art permettant d’intégrer des approches protéomiques dans le développement de leurs projets scientifiques.
ProteoSeine peut appuyer sa R&D sur les recherches actives en biologie fondamentale des équipes de l’Institut Jacques Monod, sur des collaborations avec le milieu hospitalier et sur un long savoir-faire en développement analytique de protéines thérapeutiques. L’équipe développe particulièrement son expertise dans l’analyse quantitative différentielle à haut débit de mélanges protéiques complexes, l’analyse de modifications post-traductionnelles, la caractérisation de protéines pharmaceutiques recombinantes et l’analyse de complexes non-covalents par spectrométrie de masse en conditions natives. Des approches de type bottom-up et top-down à l’échelle nano ou capillaire sont utilisées en fonction des problématiques rencontrées.
La plateforme est labellisée par le GIS IBiSA (Infrastructures pour la Biologie, la Santé et l’Agronomie) et par le Cancéropôle Ile-de-France. Elle est ouverte à l’ensemble de la communauté scientifique nationale et internationale, publique et privée. La traçabilité des activités est assurée via l’enregistrement des prestations et des utilisateurs dans un LIMS spécialement développé en interne et les données brutes sont stockées en redondance via une solution gérée globalement par l’Institut Jacques Monod.
ProteoSeine est également membre associé de ProFi, l’infrastructure nationale de recherche dédiée à l’analyse protéomique et bénéficie du soutien des organismes suivants :

ProteoSeine assure la mise en œuvre de spectromètres de masse haute résolution de type électrospray couplés à des systèmes chromatographiques à haute performance.
Les analyses bottom-up sont réalisées sur un appareil de dernière génération de type timsTOF Pro2 (Bruker) couplé à un automate de séparation haut débit (Evosep One). La technologie de mobilité ionique TIMS (Trapped Ion Mobility Spectrometry) est avant tout une technique de séparation en phase gazeuse qui résout la complexité de l’échantillon grâce à une dimension supplémentaire de séparation en plus de la chromatographie liquide à haute performance et de la spectrométrie de masse. Le dispositif TIMS accumule et concentre également les ions d’un m/z et d’une mobilité donnée. En synchronisant les cycles d’accumulation et de séquençage des ions en parallèle (technologie PASEF, Parallel Accumulation–Serial Fragmentation), l’appareil peut offrir une vitesse et une sensibilité MS/MS extrêmement élevées pour une couverture approfondie du protéome et la quantification différentielle non-biaisée de protéines.
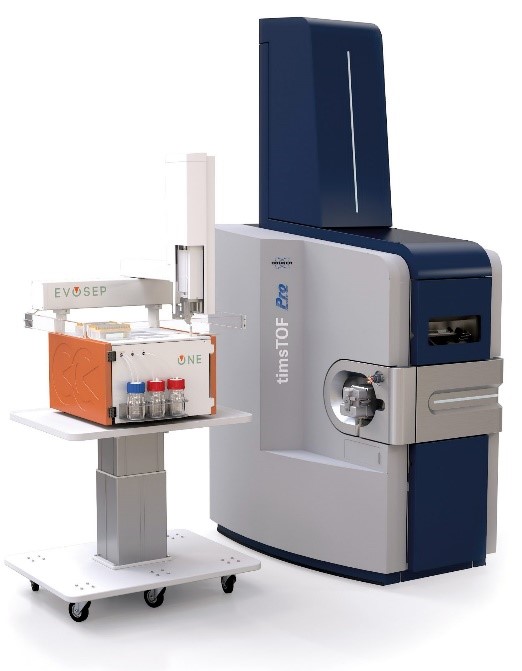
La plateforme est équipée de logiciels propriétaires permettant le retraitement des données brutes sur deux serveurs de calcul multicœurs et l’analyse statistique multivariée des résultats d’abondance. La plateforme est particulièrement en capacité de prendre en charge de larges quantités d’échantillons dans des workflows automatisés.
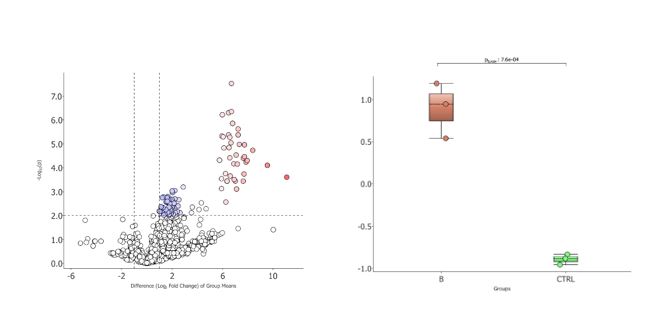
Les analyses en MS-native et sur protéine intacte sont réalisées sur un instrument de type Cyclic IMS (Waters) embarquant une technologie révolutionnaire de mobilité ionique cyclique. L’instrument est également doté de capacités de fragmentation EXD et SID en surcroît du mode de fragmentation CID disponible dans les cellules TRAP et TRANSFER. Cet instrument est couplé à une chaîne chromatographique capillaire haute pression de type M-Class ou Acquity Premier pour le développement de séparations haute résolution de protéines. Un robot nanomate ADVION est également disponible pour des analyses nanospray en infusion directe automatisée.

ProteoSeine dispose également d’une plateforme Hamilton pour la préparation automatisée d’échantillons et d’un système chromatographique haute pression pour la prise en charge de séparations de protéines suivant différentes modalités.

La plateforme assure la mise en œuvre de spectromètres de masse haute résolution de type électrospray couplés à des systèmes chromatographiques à haute performance. L’instrumentation et les outils bio-informatiques mis en place sont spécialement sélectionnés pour délivrer des résultats dans l’état de l’art de l’analyse protéomique.
La plateforme propose différents protocoles d’analyses sur des protéines intactes (top-down) ou après digestion (bottom-up) qui permettent notamment d’aborder les problématiques suivantes :
- Identification de protéines sur gel ou en solution
- Caractérisation de modifications post-traductionnelles (PTMs)
- Caractérisation de protéines recombinantes (Coverage, PTMs, process & product-related impurities, size or charge variants)
- Recherche de partenaires (Pull-Down)
- Recherche de biomarqueurs
- Analyse quantitative des variations d’abondance de protéomes (Label-Free)
- Analyse de protéomes spécifiques après enrichissement (ex : phosphoprotéome)
- Analyse de complexes par MS en conditions natives
Le principe de fonctionnement est dans un premier temps de contacter la plateforme pour définir un protocole d’analyse adapté au projet. Une fiche utilisateur précisant le fonctionnement de la plateforme sera transmise (également disponible dans la rubrique « documents »). Préalablement à toute analyse, cette fiche doit être retournée complétée avec les informations demandées. Dans un second temps, il suffit de prendre rendez-vous pour déposer les échantillons du mardi au jeudi afin de pouvoir les traiter dans les meilleurs délais.
Les résultats générés sont envoyés par e-mail dans un délai de 2 semaines en général pour des approches classiques et sont archivés. Les données brutes sont transmises à la demande car leur stockage et intégrité sont assurés par la plateforme de façon non-contractuelle. Après réception des résultats, l’utilisateur est donc responsable de l’intégrité de l’ensemble de ses données.
La tarification des prestations de la plateforme est calculée sur la base des coûts complet selon le référentiel DSFIM-SBOR-D-2014-46 du CNRS. Trois tarifications distinctes sont appliquées selon l’origine de l’émetteur du bon de commande, c’est à dire CNRS/UPC, autre académique et enfin privé. Les tarifs ci-dessous sont exprimés hors taxe ; ils sont à majorer de la TVA au taux en vigueur.
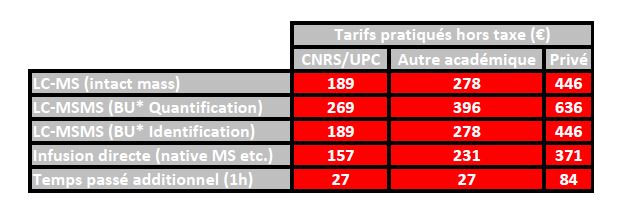
Tarification en vigueur depuis le 9 Avril 2021. * BU Bottom-Up
Les unités mixtes UPC/INSERM en délégation global de gestion INSERM listées ci-dessous bénéficient néanmoins du tarif CNRS/UPC.
| UMR | Directeur | Intitulé | Implantation |
| U942 | Alain Cohen-Solal | Biomarqueurs cardio-neuro-vasculaires : BIO-CANVAS | Hôpital Lariboisière |
| U958 | Cécile Julier | Génétique des diabètes | Faculté médecine UP |
| U965 | Marc Pocard | CARCINOSE angiogenèse et recherche transactionnelle : CART | Hôpital Lariboisière |
| U1131 | Stephane GIRAUDIER | Hématopoïèse normale et pathologique : émergence, environnement et recherche translationnelle | Hôpital Saint-Louis |
| U976 | Armand Bensussan | HIPI « Immunologie Humaine, Pathophysiologie, Immunothérapie » | Hôpital Saint-Louis |
| U1160 | Antoine Toubert | Écotaxie, micro-environnement et développement lymphocytaire | Hôpital Saint-Louis |
| U944 | Jean Soulier | Génomes, biologie cellulaire et thérapeutique | Hôpital Saint-Louis |
| U1123 | Corinne Alberti | Epidémiologie clinique et évaluation économique appliquées aux populations vulnérables : ECEVE | Faculté médecine UP |
| U1132 | Martine Cohen-Solal | Biologie de l’os et du cartilage : régulations et ciblage thérapeutique : BIOSCAR | Hôpital Lariboisière |
| U1134 | Yves Collin | Biologie intégrée du globule rouge | INTS |
| U1137 | Erik Denamur | Infection, antimicrobiens, modélisation, évolution : IAME | Faculté Bichat |
| U1141 | Pierre Gressens | Neuroprotection du cerveau en développement : PROTECT | Hôpital Robert Debré |
| U1148 | Didier Letourneur | Laboratoire de recherche vasculaire transactionnelle | Faculté Bichat |
| U1149 | Renato Monteiro | Centre de recherche sur l’inflammation : CRI | Faculté Bichat |
| U1152 | Marina Pretolani | Physiopathologie et épidémiologie des maladies respiratoires | Faculté Bichat |
| U1161 | Elisabeth Tournier-Lasserve | Génétique et physiopathologie des maladies cérébro-vasculaires | Faculté médecine UP |
L’équipe de ProteoSeine est fortement impliquée dans des actions de formation initiale et continue.
Elle dispense chaque année une semaine de formation théorique et pratique de l’analyse protéomique par spectrométrie de masse aux étudiants du M2-Pro « Ingénierie de Plateforme » d’Université Paris Cité.
Elle accueille tous les ans un étudiant de niveau M2 pour son stage pratique de fin d’études d’une durée de 6 mois.
Elle organise une formation à la spectrométrie de masse en protéomique quantitative et caractérisation de protéines recombinantes. Cette formation est référencée au catalogue de CNRS Formation Entreprise.
Elle organise 2 workshops internationaux par an dans le cadre du programme européen Twinning H2020 « MiCoBion »
Contact plateforme :
spectrodemasse@ijm.fr, +33 (0)1 57 27 81 82
Responsables
 Jean-Michel CAMADRO, Chercheur émérite, PFT/PROTEOSEINE+33 (0)1 57 27 80 95, bureau 344B
Jean-Michel CAMADRO, Chercheur émérite, PFT/PROTEOSEINE+33 (0)1 57 27 80 95, bureau 344B Guillaume CHEVREUX, Responsable plateforme ProtéoSeine, PFT/PROTEOSEINE+33 (0)1 57 27 81 19, bureau 657B
Guillaume CHEVREUX, Responsable plateforme ProtéoSeine, PFT/PROTEOSEINE+33 (0)1 57 27 81 19, bureau 657B
Membres
 Victor COCHARD, Ingénieur en analyse protéomique, PFT/PROTEOSEINE+33 (0)1 57 27 81 82, bureau 657B
Victor COCHARD, Ingénieur en analyse protéomique, PFT/PROTEOSEINE+33 (0)1 57 27 81 82, bureau 657B Veronique LEGROS, Ingénieure en protéomique, PFT/PROTEOSEINE+33 (0)1 57 27 81 82, bureau 657B
Veronique LEGROS, Ingénieure en protéomique, PFT/PROTEOSEINE+33 (0)1 57 27 81 82, bureau 657B Marie LEY, Postdoctorante, PFT/PROTEOSEINE+33 (0)1 57 27 81 19, bureau 657B
Marie LEY, Postdoctorante, PFT/PROTEOSEINE+33 (0)1 57 27 81 19, bureau 657B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
Plateforme ProteoSeine de l’Institut Jacques Monod, Bâtiment Buffon – Aile B, Pièce 657B, 6e étage – 15 rue Hélène Brion – 75205 Paris Cedex 13 – FRANCE
Charte utilisateur
4202101_Fiche Utilisateur ProteoSeine_2022
Minimiser les contaminations pour l’analyse protéomique
Minimizing contamination in Proteomics
Protocoles de lyse cellulaire pour l’analyse protéomique
Publications co-signées par les membres de la plateforme (2017-2023)
2023
Lignieres L, Sénécaut N, Dang T, Bellutti L, Hamon M, Terrier S, Legros V, Chevreux G, Lelandais G, Mège RM, Dumont J, Camadro JM. Extending the Range of SLIM-Labeling Applications: From Human Cell Lines in Culture to Caenorhabditis elegans Whole-Organism Labeling. J Proteome Res. 2023 Mar 3;22(3):996-1002. doi: 10.1021/acs.jproteome.2c00699. Epub 2023 Feb 6. PMID: 36748112; PMCID: PMC9990122.
kombo Nkoula S, Velez-Aguilera G, Ossareh-Nazari B, Van Hove L, Ayuso C, Legros V, Chevreux G, Thomas L, Seydoux G, Askjaer P, Pintard L. Mechanisms of nuclear pore complex disassembly by the mitotic Polo-like kinase 1 (PLK-1) in C. elegans embryos. Sci Adv. 2023 Jul 21;9(29):eadf7826. doi: 10.1126/sciadv.adf7826. Epub 2023 Jul 19. PMID: 37467327; PMCID: PMC10355831.
Trouillard O, Dupaigne P, Dunoyer M, Doulazmi M, Herlin MK, Frismand S, Riou A, Legros V, Chevreux G, Veaute X, Busso D, Fouquet C, Saint-Martin C, Méneret A, Trembleau A, Dusart I, Dubacq C, Roze E. Congenital mirror movements are associated with defective polymerisation of RAD51. J Med Genet. 2023 Jun 12:jmg-2023-109189. doi: 10.1136/jmg-2023-109189. PMID: 37308287.
Nashed S, El Barbry H, Benchouaia M, Dijoux-Maréchal A, Delaveau T, Ruiz-Gutierrez N, Gaulier L, Tribouillard-Tanvier D, Chevreux G, Le Crom S, Palancade B, Devaux F, Laine E, Garcia M. Functional mapping of N-terminal residues in the yeast proteome uncovers novel determinants for mitochondrial protein import. PLoS Genet. 2023 Aug 16;19(8):e1010848. doi: 10.1371/journal.pgen.1010848. PMID: 37585488.
Gareil N, Gervais A, Macaisne N, Chevreux G, Canman JC, Andreani J, Dumont J. An unconventional TOG domain is required for CLASP localization. Curr Biol. 2023 Jul 21:S0960-9822(23)00916-8. doi: 10.1016/j.cub.2023.07.009. PMID: 37516114.
Zheng Y, Cabassa-Hourton C, Eubel H, Chevreux G, Lignieres L, Crilat E, Braun HP, Lebreton S, Savouré A. Pyrroline-5-carboxylate metabolism protein complex detected in Arabidopsis thaliana leaf mitochondria. J Exp Bot. 2023 Oct 16:erad406. doi: 10.1093/jxb/erad406. PMID: 37843921.
Postic, G., Solarz, J., Loubière, C., Kandiah, J., Sawmynaden, J., Adam, F., Vilaire, M., Léger, T., Camadro, J.-M., Victorino, D. B., Potier, M.-C., Bun, E., Moroy, G., Kauskot, A., Christophe, O., & Janel, N. (2023). Over-expression of Dyrk1A affects bleeding by modulating plasma fibronectin and fibrinogen level in mice. Journal of Cellular and Molecular Medicine, n/a(n/a). https://doi.org/10.1111/jcmm.17817
Scrima N, Le Bars R, Nevers Q, Glon D, Chevreux G, Civas A, Blondel D, Lagaudriere-Gesbert C, Gaudin Y (2023) Rabies virus P protein binds to TBK1 and interferes with the formation of innate immunity-related liquid condensates. Cell Rep. 42: 111949, doi: S2211-1247(22)01850-2, PMID: 36640307
Bayot J, Martin C, Chevreux G, Camadro JM, Auchere F (2023) The adaptive response to alternative carbon sources in the pathogen Candida albicans involves a remodeling of thiol-and glutathione-dependent redox status. Biochem J. doi: BCJ20220505, PMID: 36625375
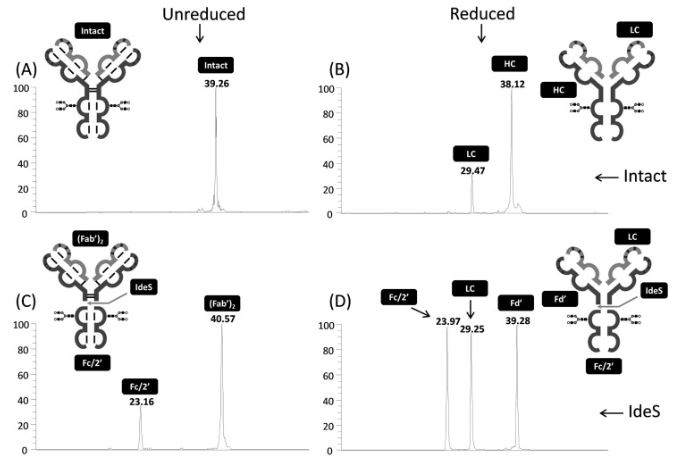
Lignieres L, Legros V, Khelil M, Senecaut N, Lauber MA, Camadro JM, Chevreux G (2023) Capillary liquid chromatography coupled with mass spectrometry for analysis of nanogram protein quantities on a wide-pore superficially porous particle column in top-down proteomics. J Chromatogr B Analyt Technol Biomed Life Sci. 1214: 123566, doi: S1570-0232(22)00471-8, PMID: 36516651
2022
Gallo G, Kotlik P, Roingeard P, Monot M, Chevreux G, Ulrich RG, Tordo N, Ermonval M (2022) Diverse susceptibilities and responses of human and rodent cells to orthohantavirus infection reveal different levels of cellular restriction. PLoS Negl Trop Dis. 16: e0010844, doi: 10.1371/journal.pntd.0010844, PMID: 36223391
Le Borgne P, Greibill L, Laporte MH, Lemullois M, Bouhouche K, Temagoult M, Rosnet O, Le Guennec M, Lignieres L, Chevreux G, Koll F, Hamel V, Guichard P, Tassin AM (2022) The evolutionary conserved proteins CEP90, FOPNL, and OFD1 recruit centriolar distal appendage proteins to initiate their assembly. PLoS Biol. 20: e3001782, doi: 10.1371/journal.pbio.3001782, PMID: 36070319
Senecaut N, Poulain P, Lignieres L, Terrier S, Legros V, Chevreux G, Lelandais G, Camadro JM (2022) Quantitative Proteomics in Yeast: From bSLIM and Proteome Discoverer Outputs to Graphical Assessment of the Significance of Protein Quantification Scores. Methods Mol Biol. 2477: 275-292, doi: 10.1007/978-1-0716-2257-5_16, PMID: 35524123
Miro-Pina C, Charmant O, Kawaguchi T, Holoch D, Michaud A, Cohen I, Humbert A, Jaszczyszyn Y, Chevreux G, Del Maestro L, Ait-Si-Ali S, Arnaiz O, Margueron R, Duharcourt S (2022) Paramecium Polycomb repressive complex 2 physically interacts with the small RNA-binding PIWI protein to repress transposable elements. Dev Cell. 57: 1037-1052 e1038, doi: 10.1016/j.devcel.2022.03.014, PMID: 35429435
Liu R, Zhang W, Gou P, Berthelet J, Nian Q, Chevreux G, Legros V, Moroy G, Bui LC, Wang L, Dupret JM, Deshayes F, Rodrigues Lima F (2022) Cisplatin causes covalent inhibition of protein-tyrosine phosphatase 1B (PTP1B) through reaction with its active site cysteine: Molecular, cellular and in vivo mice studies. Biomed Pharmacother. 153: 113372, doi: 10.1016/j.biopha.2022.113372, PMID: 35809481
Liu D, Marie JC, Pelletier AL, Song Z, Ben-Khemis M, Boudiaf K, Pintard C, Leger T, Terrier S, Chevreux G, El-Benna J, Dang PM (2022) Protein Kinase CK2 Acts as a Molecular Brake to Control NADPH Oxidase 1 Activation and Colon Inflammation. Cell Mol Gastroenterol Hepatol. 13: 1073-1093, doi: 10.1016/j.jcmgh.2022.01.003, PMID: 35031518
2021
Senecaut N, Alves G, Weisser H, Lignieres L, Terrier S, Yang-Crosson L, Poulain P, Lelandais G, Yu YK, Camadro JM (2021) Novel Insights into Quantitative Proteomics from an Innovative Bottom-Up Simple Light Isotope Metabolic (bSLIM) Labeling Data Processing Strategy. J Proteome Res. 20: 1476-1487, doi: 10.1021/acs.jproteome.0c00478, PMID: 33573382
Ribeiro-Parenti L, Jarry AC, Cavin JB, Willemetz A, Le Beyec J, Sannier A, Benadda S, Pelletier AL, Hourseau M, Leger T, Morlet B, Couvelard A, Anini Y, Msika S, Marmuse JP, Ledoux S, Le Gall M, Bado A (2021) Bariatric surgery induces a new gastric mucosa phenotype with increased functional glucagon-like peptide-1 expressing cells. Nat Commun. 12: 110, doi: 10.1038/s41467-020-20301-1, PMID: 33397977
Lemonnier T, Daldello EM, Poulhe R, Le T, Miot M, Lignieres L, Jessus C, Dupre A (2021) The M-phase regulatory phosphatase PP2A-B55delta opposes protein kinase A on Arpp19 to initiate meiotic division. Nat Commun. 12: 1837, doi: 10.1038/s41467-021-22124-0, PMID: 33758202
Lautier O, Penzo A, Rouviere JO, Chevreux G, Collet L, Loiodice I, Taddei A, Devaux F, Collart MA, Palancade B (2021) Co-translational assembly and localized translation of nucleoporins in nuclear pore complex biogenesis. Mol Cell. 81: 2417-2427 e2415, doi: 10.1016/j.molcel.2021.03.030, PMID: 33838103
Gallo G, Caignard G, Badonnel K, Chevreux G, Terrier S, Szemiel A, Roman-Sosa G, Binder F, Gu Q, Da Silva Filipe A, Ulrich RG, Kohl A, Vitour D, Tordo N, Ermonval M (2021) Interactions of Viral Proteins from Pathogenic and Low or Non-Pathogenic Orthohantaviruses with Human Type I Interferon Signaling. Viruses. 13: doi: 10.3390/v13010140, PMID: 33478127
Darrigrand R, Pierson A, Rouillon M, Renko D, Boulpicante M, Bouyssie D, Mouton-Barbosa E, Marcoux J, Garcia C, Ghosh M, Alami M, Apcher S (2021) Isoginkgetin derivative IP2 enhances the adaptive immune response against tumor antigens. Commun Biol. 4: 269, doi: 10.1038/s42003-021-01801-2, PMID: 33649389
Bertini EV, Torres MA, Leger T, Garcia C, Hong KW, Chong TM, Castellanos de Figueroa LI, Chan KG, Dessaux Y, Camadro JM, Nieto-Penalver CG (2021) Insight in the quorum sensing-driven lifestyle of the non-pathogenic Agrobacterium tumefaciens 6N2 and the interactions with the yeast Meyerozyma guilliermondii. Genomics. 113: 4352-4360, doi: 10.1016/j.ygeno.2021.11.017, PMID: 34793950
Balint B, Hergalant S, Camadro JM, Blaise S, Vanalderwiert L, Lignieres L, Gueant-Rodriguez RM, Gueant JL (2021) Fetal Programming by Methyl Donor Deficiency Produces Pathological Remodeling of the Ascending Aorta. Arterioscler Thromb Vasc Biol. 41: 1928-1941, doi: 10.1161/ATVBAHA.120.315587, PMID: 33827257
2020
Morilla I, Leger T, Marah A, Pic I, Zaag H, Ogier-Denis E (2020) Singular manifolds of proteomic drivers to model the evolution of inflammatory bowel disease status. Sci Rep. 10: 19066, doi: 10.1038/s41598-020-76011-7, PMID: 33149233
Duval C, Macabiou C, Garcia C, Lesuisse E, Camadro JM, Auchere F (2020) The adaptive response to iron involves changes in energetic strategies in the pathogen Candida albicans. Microbiologyopen. 9: e970, doi: 10.1002/mbo3.970, PMID: 31788966
2019
Scheiber IF, Pilatova J, Malych R, Kotabova E, Krijt M, Vyoral D, Mach J, Leger T, Camadro JM, Prasil O, Lesuisse E, Sutak R (2019) Copper and iron metabolism in Ostreococcus tauri – the role of phytotransferrin, plastocyanin and a chloroplast copper-transporting ATPase. Metallomics. 11: 1657-1666, doi: 10.1039/c9mt00078j, PMID: 31380866
Sanchez David RY, Combredet C, Najburg V, Millot GA, Beauclair G, Schwikowski B, Leger T, Camadro JM, Jacob Y, Bellalou J, Jouvenet N, Tangy F, Komarova AV (2019) LGP2 binds to PACT to regulate RIG-I- and MDA5-mediated antiviral responses. Sci Signal. 12: doi: 10.1126/scisignal.aar3993, PMID: 31575732
Renaudin F, Sarda S, Campillo-Gimenez L, Severac C, Leger T, Charvillat C, Rey C, Liote F, Camadro JM, Ea HK, Combes C (2019) Adsorption of Proteins on m-CPPD and Urate Crystals Inhibits Crystal-induced Cell Responses: Study on Albumin-crystal Interaction. J Funct Biomater. 10: doi: 10.3390/jfb10020018, PMID: 31027151
Nian Q, Berthelet J, Zhang W, Bui LC, Liu R, Xu X, Duval R, Ganesan S, Leger T, Chomienne C, Busi F, Guidez F, Dupret JM, Rodrigues Lima F (2019) T-Cell Protein Tyrosine Phosphatase Is Irreversibly Inhibited by Etoposide-Quinone, a Reactive Metabolite of the Chemotherapy Drug Etoposide. Mol Pharmacol. 96: 297-306, doi: 10.1124/mol.119.116319, PMID: 31221825
Massarweh A, Bosco M, Chantret I, Leger T, Jamal L, Roper DI, Dowson CG, Busca P, Bouhss A, Gravier-Pelletier C, Moore SEH (2019) Bacterial Lipid II Analogs: Novel In Vitro Substrates for Mammalian Oligosaccharyl Diphosphodolichol Diphosphatase (DLODP) Activities. Molecules. 24: doi: 10.3390/molecules24112135, PMID: 31174247
Lelandais G, Denecker T, Garcia C, Danila N, Leger T, Camadro JM (2019) Label-free quantitative proteomics in Candida yeast species: technical and biological replicates to assess data reproducibility. BMC Res Notes. 12: 470, doi: 10.1186/s13104-019-4505-8, PMID: 31370875
Le Faouder J, Gigante E, Leger T, Albuquerque M, Beaufrere A, Soubrane O, Dokmak S, Camadro JM, Cros J, Paradis V (2019) Proteomic Landscape of Cholangiocarcinomas Reveals Three Different Subgroups According to Their Localization and the Aspect of Non-Tumor Liver. Proteomics Clin Appl. 13: e1800128, doi: 10.1002/prca.201800128, PMID: 30520266
Kundlacz C, Pourcelot M, Fablet A, Amaral Da Silva Moraes R, Leger T, Morlet B, Viarouge C, Sailleau C, Turpaud M, Gorlier A, Breard E, Lecollinet S, van Rijn PA, Zientara S, Vitour D, Caignard G (2019) Novel Function of Bluetongue Virus NS3 Protein in Regulation of the MAPK/ERK Signaling Pathway. J Virol. 93: doi: 10.1128/JVI.00336-19, PMID: 31167915
Fernandez J, Machado AK, Lyonnais S, Chamontin C, Gartner K, Leger T, Henriquet C, Garcia C, Portilho DM, Pugniere M, Chaloin L, Muriaux D, Yamauchi Y, Blaise M, Nisole S, Arhel NJ (2019) Transportin-1 binds to the HIV-1 capsid via a nuclear localization signal and triggers uncoating. Nat Microbiol. 4: 1840-1850, doi: 10.1038/s41564-019-0575-6, PMID: 31611641
Elzaiat M, Herman L, Legois B, Leger T, Todeschini AL, Veitia RA (2019) High-throughput Exploration of the Network Dependent on AKT1 in Mouse Ovarian Granulosa Cells. Mol Cell Proteomics. 18: 1307-1319, doi: 10.1074/mcp.RA119.0014613, PMID: 30992313
El Banna N, Hatem E, Heneman-Masurel A, Leger T, Baille D, Vernis L, Garcia C, Martineau S, Dupuy C, Vagner S, Camadro JM, Huang ME (2019) Redox modifications of cysteine-containing proteins, cell cycle arrest and translation inhibition: Involvement in vitamin C-induced breast cancer cell death. Redox Biol. 26: 101290, doi: 10.1016/j.redox.2019.101290, PMID: 31412312
Bouillier C, Cosentino G, Leger T, Rincheval V, Richard CA, Desquesnes A, Sitterlin D, Blouquit-Laye S, Eleouet JF, Gault E, Rameix-Welti MA (2019) The Interactome analysis of the Respiratory Syncytial Virus protein M2-1 suggests a new role in viral mRNA metabolism post-transcription. Sci Rep. 9: 15258, doi: 10.1038/s41598-019-51746-0, PMID: 31649314
Bonnet J, Garcia C, Leger T, Couquet MP, Vignoles P, Vatunga G, Ndung’u J, Boudot C, Bisser S, Courtioux B (2019) Proteome characterization in various biological fluids of Trypanosoma brucei gambiense-infected subjects. J Proteomics. 196: 150-161, doi: 10.1016/j.jprot.2018.11.005, PMID: 30414516
2018
Telot L, Rousseau E, Lesuisse E, Garcia C, Morlet B, Leger T, Camadro JM, Serre V (2018) Quantitative proteomics in Friedreich’s ataxia B-lymphocytes: A valuable approach to decipher the biochemical events responsible for pathogenesis. Biochim Biophys Acta Mol Basis Dis. 1864: 997-1009, doi: 10.1016/j.bbadis.2018.01.010, PMID: 29329987
Obino D, Fetler L, Soza A, Malbec O, Saez JJ, Labarca M, Oyanadel C, Del Valle Batalla F, Goles N, Chikina A, Lankar D, Segovia-Miranda F, Garcia C, Leger T, Gonzalez A, Espeli M, Lennon-Dumenil AM, Yuseff MI (2018) Galectin-8 Favors the Presentation of Surface-Tethered Antigens by Stabilizing the B Cell Immune Synapse. Cell Rep. 25: 3110-3122 e3116, doi: 10.1016/j.celrep.2018.11.052, PMID: 30540943
Denard J, Rouillon J, Leger T, Garcia C, Lambert MP, Griffith G, Jenny C, Camadro JM, Garcia L, Svinartchouk F (2018) AAV-8 and AAV-9 Vectors Cooperate with Serum Proteins Differently Than AAV-1 and AAV-6. Mol Ther Methods Clin Dev. 10: 291-302, doi: 10.1016/j.omtm.2018.08.001, PMID: 30155509
2017
Richarme G, Liu C, Mihoub M, Abdallah J, Leger T, Joly N, Liebart JC, Jurkunas UV, Nadal M, Bouloc P, Dairou J, Lamouri A (2017) Guanine glycation repair by DJ-1/Park7 and its bacterial homologs. Science. 357: 208-211, doi: 10.1126/science.aag1095, PMID: 28596309
Planchon M, Leger T, Spalla O, Huber G, Ferrari R (2017) Metabolomic and proteomic investigations of impacts of titanium dioxide nanoparticles on Escherichia coli. PLoS One. 12: e0178437, doi: 10.1371/journal.pone.0178437, PMID: 28570583
Leger T, Garcia C, Collomb L, Camadro JM (2017) A Simple Light Isotope Metabolic Labeling (SLIM-labeling) Strategy: A Powerful Tool to Address the Dynamics of Proteome Variations In Vivo. Mol Cell Proteomics. 16: 2017-2031, doi: 10.1074/mcp.M117.066936, PMID: 28821603
Hovsepian J, Defenouillere Q, Albanese V, Vachova L, Garcia C, Palkova Z, Leon S (2017) Multilevel regulation of an alpha-arrestin by glucose depletion controls hexose transporter endocytosis. J Cell Biol. 216: 1811-1831, doi: 10.1083/jcb.201610094, PMID: 28468835
Gergondey R, Garcia C, Marchand CH, Lemaire SD, Camadro JM, Auchere F (2017) Modulation of the specific glutathionylation of mitochondrial proteins in the yeast Saccharomyces cerevisiae under basal and stress conditions. Biochem J. 474: 1175-1193, doi: 10.1042/BCJ20160927, PMID: 28167699
Boukpessi T, Hoac B, Coyac BR, Leger T, Garcia C, Wicart P, Whyte MP, Glorieux FH, Linglart A, Chaussain C, McKee MD (2017) Osteopontin and the dento-osseous pathobiology of X-linked hypophosphatemia. Bone. 95: 151-161, doi: 10.1016/j.bone.2016.11.019, PMID: 27884786
15/06/2023 :
La plateforme ProtéoSeine organise un séminaire conjointement avec la société Protein Metrics. Ce séminaire aura pour but de présenter le travail effectué par la plateforme, notamment sur l’analyse de protéines intactes.
La société Protein Metrics présentera quant à elle ses différentes solutions logicielles, pour l’analyse de protéines, peptides et oligonucléotides.
Ce séminaire aura lieu dans la matinée du jeudi 15 juin 2023, à l’Institut Jacques Monod.
Envie de participer ?
Vous pouvez visionner le programme et vous inscrire ici : https://proteinmetrics.com/paris-symposium-2023/
06/02/2023
L’équipe de Jean-Michel Camadro en collaboration avec la plateforme ProtéoSeine, l’équipe Ladoux/Mège et Dumont viennent de publier un nouvel article :
Laurent Lignières, Nicolas Sénécaut, Tien Dang, Laura Bellutti, Marion Hamon, Samuel Terrier, Véronique Legros, Guillaume Chevreux, Gaëlle Lelandais, René-Marc Mège, Julien Dumont, and Jean-Michel Camadro, Journal of Proteome Research, 2023, 22, 3, 996–1002
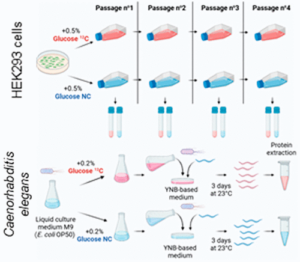
07/02/2023
L’équipe de Jean-Michel Camadro en collaboration avec la plateforme ProtéoSeine viennent de publier un nouvel article :
Juliette Bayot, Caroline Martin, Guillaume Chevreux, Jean-Michel Camadro, Françoise Auchère, Biochemical Journal Volume 480, Issue 3.
05/01/2023 : la plateforme Protoseine vient de publier un nouvel article dans Journal of Chromatography B :
29/11/2022 : Inauguration de nouveaux équipements lors des 40 ans de l’IJM avec la Région Ile-de-France