Spécification des destins cellulaires
Jérôme COLLIGNON
Nous étudions le rôle d’une protéine sécrétée, appelée NODAL, dans la spécification des lignages cellulaires au cours de l’embryogénèse. Cette thématique nous a conduit à nous intéresser aussi au rôle de cette molécule dans la progression tumorale.
Mots-clés : Embryon de souris, cellules souches embryonnaires, mélanome humain, signalisation ACTIVINE/NODAL, régulation de l’expression génétique, enhancers, pluripotence.
+33 (0)1 57 27 81 08 Contact
Nous étudions le rôle d’une protéine sécrétée, appelée NODAL, dans la spécification des lignages cellulaires au cours de l’embryogénèse. Cette thématique nous a conduit à nous intéresser aussi au rôle de cette molécule dans la progression tumorale.
Les études menées chez l’embryon de souris ont montré que la spécification des destins cellulaires implique plusieurs voies de signalisation. Ces voies participent aussi à la maintenance et au renouveau des tissus chez l’adulte, des fonctions qui sont en causes dans de nombreuses pathologies. L’étude de leur implication et de leurs interactions pendant le développement embryonnaire, lorsque l’organisme est encore relativement simple, est donc particulièrement pertinente pour comprendre leur rôle plus tard, dans des contextes plus complexes.
NODAL est un membre de la famille des TGFß. Son signal est transmis par ce que l’on appelle la voie de signalisation ACTIVINE/NODAL, qui dépend des facteurs cytoplasmiques SMAD2 et 3. Le gène Nodal est requis pour la mise en place des axes embryonnaires. Il est aussi requis pour la spécification et la maintenance de diverses identités cellulaires, aussi bien embryonnaires qu’extra-embryonnaires. Plusieurs de ces types cellulaires ont en commun d’être motiles. Le locus Nodal a été l’objet de nombreuses études, ses séquences régulatrices ont été identifiées et caractérisées, et c’est devenu un modèle pour comprendre comment diverses influences (signaux TGFß, WNT et NOTCH, facteurs de pluripotence, etc.) sont intégrées et font qu’un gène est exprimé dans une certaine cellule à un moment donné. Chez l’adulte Nodal est exprimé dans des tissus qui sont renouvelés ou remodelés de façon périodique sous le contrôle de stimuli hormonaux, tels l’endomètre et la glande mammaire. Chez l’homme, l’expression de NODAL a été détectée dans les cellules tumorales de différents types de cancers, et elle a été associée à leur plasticité et à leur comportement invasif, suggérant que son rôle dans ce contexte reflète ce que l’on sait de ses fonctions dans les tissus adultes et embryonnaires.
Nous employons actuellement une combinaison d’approches de génétique, de biologie moléculaire, de transcriptomique et d’imagerie pour étudier le rôle de NODAL pendant la spécification et la différenciation des premiers lignages cellulaires, aussi bien chez l’embryon de souris que dans des modèles de culture in vitro reposant sur l’utilisation de cellules souches embryonnaires. L’étude de ces modèles in vitro est réalisée en collaboration avec des biophysiciens qui apportent leur expertise en microfluidique, traitement de données quantitatives et modélisation. Enfin, nous utilisons des approches de biologie cellulaire, de génétique, de biochimie, de transcriptomique et de spectrométrie de masse pour étudier les mécanismes par lesquels un gène antisens du gène NODAL promeut la progression tumorale dans un modèle cellulaire de mélanome humain.
Étude du rôle du gène Nodal au cours du développement préimplantatoire
L’expression de Nodal est détectée pour la première fois dans la masse cellulaire interne (MCI) du blastocyste au jour embryonnaire 3,5 (E3.5) (Granier et al., 2011 ; Takaoka et al., 2006 ; Takaoka et al., 2017). À E3.75, lorsque la MCI donne naissance à l’épiblaste pluripotent (Epi) et à l’endoderme primitif (PrE), un tissu extra-embryonnaire, l’expression de Nodal persiste dans l’Epi mais devient plus forte dans les cellules du PrE. A E4.5, lorsque l’embryon s’implante, l’expression de Nodal est diminuée dans le PrE mais augmente dans l’Epi. On a constaté que l’activité de la voie de signalisation ACTIVINE/NODAL à ces stades suit de près l’expression de Nodal (Granier et al., 2011).
Le défaut le plus précoce décrit chez les embryons Nodal-/- jusqu’à présent est leur incapacité à activer l’expression de Lefty1, un antagoniste de NODAL qui est également une cible de sa voie de signalisation, dans les cellules de la MCI et du PrE entre E3.5 et E4.5 (Takaoka et al., 2017). Les embryons E4,5 Nodal-/- comprennent néanmoins les trois couches cellulaires qui constituent normalement un blastocyste en cours d’implantation. Ce n’est qu’après l’implantation que leurs défauts de développement caractéristiques, l’incapacité à établir des identités cellulaires embryonnaires et extra-embryonnaires spécifiques, à établir un axe AP et à gastruler, ont été caractérisés (Robertson 2014). Outre Lefty1, d’autres cibles possibles de NODAL aux stades préimplantatoires ont été identifiées dans des études sur les cellules souches embryonnaires (ESCs), qui sont représentatives des cellules Epi précoces (Galvin et al., 2010; Mullen et al., 2011). Compte tenu de la prééminence de certaines de ces cibles, qui comprennent des facteurs de transcription et des composants des voies de signalisation, l’absence d’un phénotype préimplantatoire peut sembler surprenante. Une raison possible de cette absence apparente de défauts précoces est la présence à ces stades de ligands d’origine zygotique (ACTIVINE) et utérine (ACTIVINE, NODAL) susceptibles de compenser l’absence de NODAL zygotique (Papanayotou & Collignon, 2014). Le fait que la signalisation ACTIVINE/NODAL est active dans l’embryon bien avant que l’expression de Nodal zygotique ne commence, est en accord avec cette possibilité (James et al., 2005).
Afin de révéler les contributions respectives de NODAL et d’ACTIVINE à la formation du blastocyste nous réalisons donc actuellement une comparaison des phénotypes d’embryons exposés à divers traitements inducteurs ou inhibiteurs avec ceux d’embryons Nodal-/- fraîchement disséqués ou cultivés ex-utero.
Ce programme a été soutenu par le programme Émergence (IDEX Université Paris Cité), la Fondation ARC, l’École Doctorale BioSPC, l’EUR G.E.N.E et le Labex Who AM I?

Etude du mécanisme de la régionalisation de la ligne primitive
Nous cherchons à comprendre comment la ligne primitive (LP), dont la formation marque le début de la gastrulation, est régionalisée chez l’embryon de souris. La LP apparaît au sein de l’épiblaste proximal postérieur, et s’étend progressivement vers son extrémité distale. Les cellules émergeant précocement de la région proximale/postérieure de la LP vont migrer de manière proximale pour former le mésoderme extra-embryonnaire ou de manière latérale pour former le mésoderme embryonnaire, tandis que les cellules émergeant de sa région distale/antérieure vont former le mésoderme axial et l’endoderme définitif. Des mutations de perte de fonction complètes et partielles ont montré que trois protéines sécrétées, WNT3, BMP4 et NODAL, sont essentielles à la formation et à la régionalisation de la LP. Ces études ont abouti à un modèle dans lequel des gradients opposés de signalisation de NODAL et de BMP4 régissent l’allocation des destins cellulaires de la LP, la signalisation BMP la plus élevée favorisant le mésoderme extra-embryonnaire dans la LP postérieure, et la signalisation NODAL la plus élevée favorisant l’endoderme définitif dans la LP antérieure (Tam et al., 2006 ; Arnold & Robertson 2007). S’il y a bien des indications qu’un tel gradient de signalisation BMP existe (Yamamoto et al., 2009), c’est beaucoup moins clair pour la signalisation NODAL (Camacho-Aguilar & Warmflash, 2020). Des résultats obtenus chez le poisson zèbre suggèrent qu’une longue exposition à la signalisation NODAL favorise effectivement les destins LP antérieurs (Hill, 2018), mais dans l’embryon de souris, il a été difficile d’établir un lien entre exposition au ligand et destin cellulaire. En outre, l’interdépendance des expressions de Wnt3, Bmp4 et Nodal, et leurs rôles dans la régionalisation de l’embryon à des stades plus précoces rendent difficile de séparer, par exemple, l’effet du manque de NODAL de ceux d’un manque de BMP4 ou de WNT3 dans les défauts de régionalisation des embryons Nodal-/-. Enfin, la faible quantité de matériel biologique dans un embryon rend difficile l’application de méthodes biochimiques pour étudier comment les décisions relatives à la détermination de l’identité cellulaire intègrent ces 3 signaux. Cependant, le développement récent de méthodes qui utilisent des cellules pluripotentes pour récapituler la gastrulation in vitro, permettant ainsi un meilleur contrôle des conditions dans lesquelles la régionalisation a lieu, rend maintenant possibles ces investigations.
Afin de comparer les événements qui se déroulent in vitro avec ceux qui se déroulent réellement dans l’embryon de souris, notre collaborateur Benoit Sorre (UMR168 Institut Curie/CNRS) a adapté une méthode qu’il avait initialement développée avec des cellules souches embryonnaires humaines (hESCs) (Warmflash et al., 2014) à l’utilisation de cellules souches pluripotentes de souris représentatives de l’épiblaste pré-gastrulation, les EpiLCs (Plouhinec et al., 2021). Ce modèle in vitro de gastrulation en 2D, ou gastruloïde 2D de souris (m2DGas), présente de nombreux avantages. Il est simple à mettre en oeuvre, plusieurs colonies peuvent être générées en même temps, et il se prête à l’imagerie et à la quantification. Nous avons constaté, comme d’autres (Morgani et al., 2018), que les m2DGas, s’auto-organisent de manière reproductible lorsqu’ils sont exposés à BMP4, formant en 48H une LP qui donne ensuite naissance à un anneau de cellules mésodermiques entourant un noyau d’ectoderme. Nous utilisons maintenant un ensemble d’approches (Immunofluorescence, hybridation in situ, RT-qPCR, RNAseq en cellules uniques, inférence de réseaux de régulation, essai de ligature de proximité) pour étudier dans ce modèle le mécanisme qui sous-tend la régionalisation de la LP.
Ce projet a été soutenu par l’INCa et l’École Doctorale BioSPC. Il est actuellement soutenu par l’ANR.
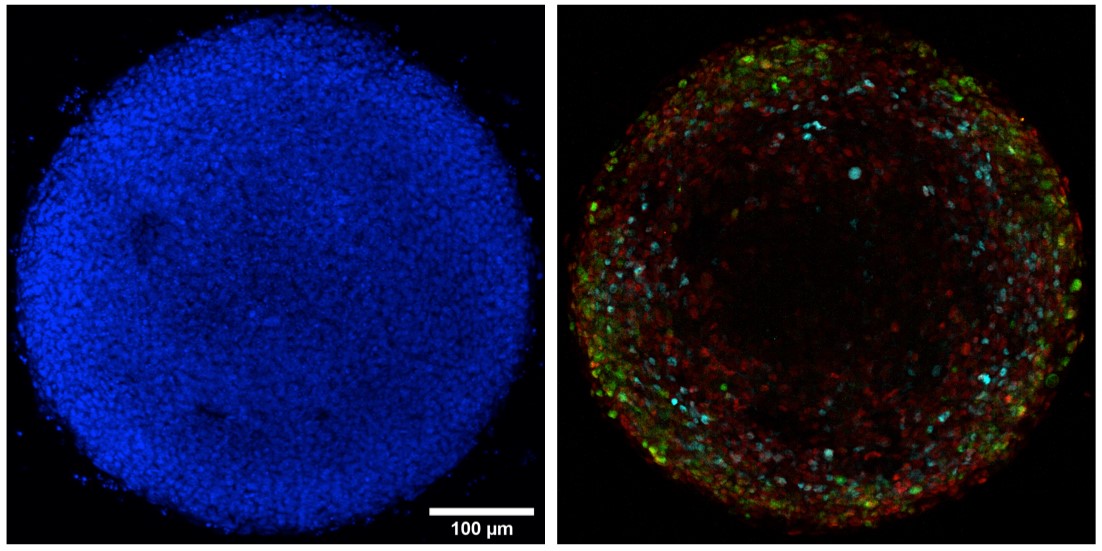
Colonie EpiLC de 500 mm de diamètre après stimulation BMP4 pendant 36h. A gauche, le marquage des noyaux au Hoechst (bleu) permet de visualiser l’ensemble des cellules, à droite le marquage par immunofluorescence permet d’identifier les cellules de la ligne primitive et du mésoderme naissant (BRA en rouge), du mésoderme extra-embryonnaires (CDX2 en vert) et de la ligne primitive proximale (FOXA2 en cyan).
Étude du rôle du gène NODAL, antisens de LADON, dans la progression tumorale du mélanome humain
L’implication de NODAL dans la progression tumorale et les métastases a été décrite pour la première fois dans des lignées cellulaires de mélanome (Topczewska et al., 2006). Cependant, alors que des preuves soutenant un tel rôle pour ce ligand ont été obtenues dans d’autres types de cancer, sa contribution réelle à la formation de métastases dans le mélanome a été contestée (Donovan et al., 2017; Hooijkaas et al., 2011), notamment parce que son expression n’était pas toujours détectée. Une revue des données disponibles sur l’expression de NODAL dans les cellules cancéreuses a suggéré l’existence de variants d’épissage (Strizzi et al., 2012), incluant tous le deuxième exon du gène mais dépourvus parfois, notamment dans les lignées cellulaires de mélanome, de son troisième (et dernier) exon et donc de la capacité à produire un ligand fonctionnel. Cette observation fournit une explication aux différences constatées entre les niveaux d’expression de NODAL rapportés par différentes études pour ces lignées cellulaires, mais soulève également la possibilité d’une fonction du gène indépendante de la protéine qu’il encode.
Nous avons donc utilisé l’édition du génome pour supprimer l’exon2 de NODAL dans la lignée cellulaire de mélanome A375, un modèle cellulaire reconnu de mélanome humain précédemment décrit comme exprimant NODAL (Topczewska et al., 2006). Cette mutation a réduit de manière drastique la capacité de ces cellules à acquérir un phénotype invasif. Cependant, l’inhibition ou la stimulation exogène de la signalisation ACTIVINE/NODAL n’a eu aucun effet sur le comportement des cellules A375, et nous n’avons pas pu détecter l’expression de NODAL dans ces cellules, ce qui implique que ni le ligand NODAL ni son ARNm ne jouent un rôle dans ce phénotype. Nous avons cependant découvert que la mutation désactivait l’expression d’un transcrit antisens naturel de 1750 nucléotides de long, chevauchant l’exon2 entier de NODAL (Fig.1). Identifié à l’origine dans le glioblastome, il était jusqu’à présent passé largement inaperçu. Nous avons appelé ce transcrit LADON et confirmé qu’il s’agit bien de ce que des études antérieures avaient identifié par erreur comme étant l’expression de NODAL dans les lignées cellulaires de mélanome, ainsi que dans certaines autres lignées cellulaires cancéreuses. L’expression forcée de LADON dans des cellules A375 mutantes pour l’exon2 de NODAL a permis de sauver les défauts constatés dans ces cellules, et a donc confirmé son implication dans l’acquisition d’un comportement invasif.
Il existe de multiples exemples de l’implication des longs ARN non codants (lncRNA) dans la physiologie des tissus et dans la dérégulation des processus cellulaires qui aboutissent au cancer (Huarte 2015 ; Iyer et al., 2015 ; Schmitt & Chang). C’est la raison de l’intérêt que suscitent actuellement les lncRNA en tant que biomarqueurs pour le diagnostic et le pronostic, mais aussi en tant que cibles potentielles pour la thérapie ou en tant qu’outils pour corriger l’expression génique défectueuse qui sous-tend le cancer et d’autres pathologies (Wahlestedt 2013 ; Slaby et al., 2017). Bien que nous ayons caractérisé certains des événements qui dépendent de l’expression de LADON dans les cellules de mélanome, nous ne savons pas comment il les déclenche réellement. Nos objectifs sont donc d’identifier les cibles directes de LADON et de caractériser la manière dont il contrôle leur expression.
Ce projet a été soutenu par l’INCa et le GEFLUC.
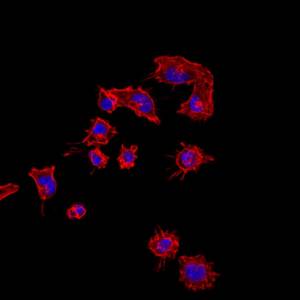
Cellules de mélanome de la lignée A375, dont l’acquisition d’un comportement invasif dépend de l’expression du transcrit LADON. Les noyaux sont en bleu et le cytosquelette en rouge.
Membres
 Nazareno BISSON, Stagiaire, COLLIGNON LAB
Nazareno BISSON, Stagiaire, COLLIGNON LAB Annie DUTRIAUX, Ingénieure en biologie, COLLIGNON LAB+33 (0)1 57 27 81 10, bureau 502B
Annie DUTRIAUX, Ingénieure en biologie, COLLIGNON LAB+33 (0)1 57 27 81 10, bureau 502B Domenico FLAGIELLO, Enseignant-chercheur, COLLIGNON LAB+33 (0)1 57 27 80 86, bureau 502B
Domenico FLAGIELLO, Enseignant-chercheur, COLLIGNON LAB+33 (0)1 57 27 80 86, bureau 502B Futura GATTOBIGIO, Doctorante, COLLIGNON LAB+33 (0)1 57 27 81 10, bureau 502B
Futura GATTOBIGIO, Doctorante, COLLIGNON LAB+33 (0)1 57 27 81 10, bureau 502B Samra KECHEMIR, Stagiaire, COLLIGNON LAB
Samra KECHEMIR, Stagiaire, COLLIGNON LAB Mikel RELLOSO ORTIZ DE URIARTE, Stagiaire, COLLIGNON LAB
Mikel RELLOSO ORTIZ DE URIARTE, Stagiaire, COLLIGNON LAB Gaël SIMON, Postdoctorant, COLLIGNON LAB+33 (0)1 57 27 81 09, bureau 502B
Gaël SIMON, Postdoctorant, COLLIGNON LAB+33 (0)1 57 27 81 09, bureau 502B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
BMP signaling modulations control primitive streak patterning. Simon G, Plouhinec JL, Gilardi-Hebenstreit P, Sorre B*, Collignon J*. bioRxiv 2024.10.01.616050; doi: https://doi.org/10.1101/2024.10.01.616050
LADON, a natural antisense transcript of NODAL, promotes metastasis promotes tumour progression and metastasis in melanoma. Dutriaux A, Diazzi S, Bresesti C, Hardouin S, Deshayes F, Collignon J*, Flagiello D*. Non-Coding RNA. 2023 Nov 15;9(6):71. doi: 10.3390/ncrna9060071. PMID: 37987367
Dissecting signalling hierarchies in the patterning of the mouse primitive streak using micro-patterned EpiLC colonies. Plouhinec JL#, Simon G#, Vieira M, Collignon J*, Sorre B*. Stem Cell reports. 2022 Jul 12;17(7):1757-1771. doi: 10.1016/j.stemcr.2022.05.009. PMID: 35714597
Notch activation interferes with cell fate specification in the gastrulating mouse embryo. Souilhol C#, Perea-Gomez A#, Camus A, Beck-Cormier S, Vandormael-Pournin S, Escande M, Collignon J and Cohen-Tannoudji M. Development. 2015 Nov 1;142(21):3649-60. doi: 10.1242/dev.121145. PMID: 26534985
A conserved role for non-neural ectoderm cells in early neural development. Cajal M, Creuzet S, Papanayotou C, Sabéran-Djoneidi D, Chuva de Sousa Lopes SM, Zwijsen A, Collignon J and Camus A. Development. 2014 Nov;141(21):4127-38. doi: 10.1242/dev.107425. Epub 2014 Oct 1. PMID: 25273086
Activin/Nodal signalling before implantation: setting the stage for embryo patterning. Papanayotou C and Collignon J. Philos Trans R Soc Lond B Biol Sci. 2014 Dec 5;369(1657):20130539. doi: 10.1098/rstb.2013.0539. PMID: 25349448
A novel Nodal enhancer dependent on pluripotency factors and Smad2/3 signaling conditions a regulatory switch during epiblast maturation. Papanayotou C, Benhaddou A, Camus A, Perea-Gomez A, Jouneau A, Mezger V, Langa F, Ott S, Sabéran-Djoneidi D, Collignon J. PLoS Biol. 2014 Jun 24;12(6):e1001890. doi: 10.1371/journal.pbio.1001890. eCollection 2014 Jun. PMID: 24960041
Clonal and molecular analysis of the prospective anterior neural boundary in the mouse embryo. Cajal M, Lawson KA, Hill B, Moreau A, Rao J, Ross A, Collignon J, Camus A. Development. 2012 Jan;139(2):423-36. doi: 10.1242/dev.075499. PMID: 22186731
Nodal cis-regulatory elements reveal epiblast and primitive endoderm heterogeneity in the peri-implantation mouse embryo. Granier C#, Gurchenkov V#, Perea-Gomez A, Camus A, Ott S, Papanayotou C, Iranzo J, Moreau A, Reid J, Koentges G, Sabéran-Djoneidi D, Collignon J. Dev Biol. 2011 Jan 15;349(2):350-62. doi: 10.1016/j.ydbio.2010.10.036. Epub 2010 Nov 1. PMID: 21047506
Absence of Nodal signalling promotes precocious neural differentiation in the mouse embryo. Camus A, Perea-Gomez A, Moreau A and Collignon J. Dev Biol. 2006 Jul 15;295(2):743-55. doi: 10.1016/j.ydbio.2006.03.047. Epub 2006 Apr 7. PMID: 16678814
Initiation of gastrulation in the mouse embryo is preceded by an apparent shift in the orientation of the anterior-posterior axis. Perea-Gomez A, Camus A, Moreau A, Grieve K, Moneron G, Dubois A, Cibert C and Collignon J. Curr Biol. 2004 Feb 3;14(3):197-207. doi: 10.1016/j.cub.2004.01.030. PMID: 14761651BMP signaling modulations control primitive streak patterning. Simon G, Plouhinec JL, Gilardi-Hebenstreit P, Sorre B*, Collignon J*. bioRxiv 2024.10.01.616050; doi: https://doi.org/10.1101/2024.10.01.616050
LADON, a natural antisense transcript of NODAL, promotes metastasis promotes tumour progression and metastasis in melanoma. Dutriaux A, Diazzi S, Bresesti C, Hardouin S, Deshayes F, Collignon J*, Flagiello D*. Non-Coding RNA. 2023 Nov 15;9(6):71. doi: 10.3390/ncrna9060071. PMID: 37987367
Addressing Critical Issues Related to Storage and Stability of the Vault Nanoparticle Expressed and Purified from Komagataella phaffi. Tomaino G, Pantaleoni C, Ami D, Pellecchia F, Dutriaux A, Barbieri L, Garbujo S, Natalello A, Tortora P, Frascotti G. Int J Mol Sci. 2023 Feb 20;24(4):4214. doi: 10.3390/ijms24044214. PMID: 36835627
Dissecting signalling hierarchies in the patterning of the mouse primitive streak using micro-patterned EpiLC colonies. Plouhinec JL#, Simon G#, Vieira M, Collignon J*, Sorre B*. Stem Cell reports. 2022 Jul 12;17(7):1757-1771. doi: 10.1016/j.stemcr.2022.05.009. PMID: 35714597
Whole embryo culture, transcriptomics and RNA interference identify TBX1 and FGF11 as novel regulators of limb development in the mouse. Tejedor G, Laplace-Builhé B, Luz-Crawford P, Assou S, Barthelaix A, Mathieu M, Kissa K, Jorgensen C, Collignon J*, Chuchana P*, Djouad F*. Sci Rep. 2020 Feb 27;10(1):3597. doi: 10.1038/s41598-020-60217-w. PMID: 32107392
Loss of Cubilin, the intrinsic factor-vitamin B12 receptor, impairs visceral endoderm endocytosis and endodermal patterning in the mouse. Perea-Gomez A, Cases O, Lelièvre V, Pulina MV, Collignon J, Hadjantonakis AK, Kozyraki R. Sci Rep. 2019 Jul 15;9(1):10168. doi: 10.1038/s41598-019-46559-0. PMID: 31308417
Evolutionary Gain of Dbx1 Expression Drives Subplate Identity in the Cerebral Cortex. Arai Y, Cwetsch AW, Coppola E, Cipriani S, Nishihara H, Kanki H, Saillour Y, Freret-Hodara B, Dutriaux A, Okada N, Okano H, Dehay C, Nardelli J, Gressens P, Shimogori T, D’Onofrio G, Pierani A. Cell Rep. 2019 Oct 15;29(3):645-658.e5. doi: 10.1016/j.celrep.2019.09.007. PMID: 31618633
A requirement for ZIC2 in the regulation of Nodal expression underlies the establishment of left-sided identity. Dykes IM, Szumska D, Kuncheria L, Puliyadi R, Chen CM, Papanayotou C, Lockstone H, Dubourg C, David V, Schneider JE, Keane TM, Adams DJ, Brown SDM, Mercier S, Odent S, Collignon J, Bhattacharya S. (2018). Sci Rep. 2018 Jul 11;8(1):10439. doi: 10.1038/s41598-018-28714-1. PMID: 29992973
Anteroposterior polarity and elongation in the absence of extraembryonic tissues and spatially localised signalling in Gastruloids, mammalian embryonic organoids. Turner DA, Girgin M, Alonso-Crisostomo L, Trivedi V, Baillie-Johnson P, Glodowski CR, Hayward PC, Collignon J, Gustavsen C, Serup P, Steventon B, Lutolf M, and Martinez Arias A. Development. 2017 Nov 1;144(21):3894-3906. doi: 10.1242/dev.150391. Epub 2017 Sep 26. PMID: 28951435
Paternal Insulin-like Growth Factor 2 (Igf2) Regulates Stem Cell Activity During Adulthood. Barroca V, Lewandowski D, Jaracz-Ros A, Hardouin SN. EBioMedicine. 2017 Feb;15:150-162. doi: 10.1016/j.ebiom.2016.11.035. Epub 2016 Dec 3. PMID: 28007480
An evolutionary, structural and functional overview of the mammalian TEAD1 and TEAD2 transcription factors. Landin-Malt A, Benhaddou A, Zider A, Flagiello D. Gene. 2016 Oct 10;591(1):292-303. doi: 10.1016/j.gene.2016.07.028. Epub 2016 Jul 14. PMID: 27421669
Notch activation interferes with cell fate specification in the gastrulating mouse embryo. Souilhol C#, Perea-Gomez A#, Camus A, Beck-Cormier S, Vandormael-Pournin S, Escande M, Collignon J and Cohen-Tannoudji M. Development. 2015 Nov 1;142(21):3649-60. doi: 10.1242/dev.121145. PMID: 26534985
- Futura Gattobigio (en cours) – Université Paris Cité : Mechanism and role of NODAL signalling at preimplantation stages
- Giulia Tomaino (2024) – University of Milano-Bicocca (cotutelle/Dept of Biotechnology and Biosciences) : Leveraging recombinant Vault nanoparticles produced in Komagataella phaffii for targeted delivery of siRNAs as therapeutic molecules
- Gaël Simon (2023) – Université Paris Cité : In vitro study of BMP, WNT and NODAL signalling pathways during primitive streak formation and specification using mouse 2D Gastruloids
- Nicolas Porchet (2019) – Université de Paris : Role of signaling pathways in cell-fate specification in the early mouse embryo
- Mathieu Vieira (2018) – Université Paris Diderot : Régulation de l’expression du gène Nodal lors de la différenciation des cellules souches embryonnaires
- Marieke Cajal (2013) – Université Paris Diderot : Étude des étapes précoces de la formation et de la régionalisation du cerveau des vertébrés : Un rôle conservé des cellules de l’ectoderme non-neural chez la souris et le poulet
- Elsa Mazari (2013) – Université Paris Sud (cotutelle/Laboratoire de Photonique et de Nanostructures, LPN-CNRS, Marcoussis) : Microsystèmes magnétiques et électriques pour la modification spatio-temporelle de voies de signalisation biologiques
- Vasily Gurchenkov (2009) – Université Pierre et Marie Curie : Functional study of the asymmetric specific element of the Nodal gene during early mouse development
- Céline Granier (2007) – Université Paris Diderot : Étude de la dynamique de la voie de signalisation NODAL chez l’embryon de souris aux stades péri-implantatoires
Bertrand Cosson, EDC, Paris, France
- Gabriella MINCHIOTTI, IGB, Naples, Italie
- Claire Chazaud, Institut GReD, Clermont-Ferrand, France
- Jean-Léon MAITRE, Institut Curie, Paris, France
- Caroline HILL, Crick Institute, Londres, Grande-Bretagne
- Juliette AZIMZADEH, Institut Jacques Monod, Paris, France
- Benoit SORRE, Institut Curie, Paris, France
- Hervé ISAMBERT, Institut Curie, Paris, France
- Sandrine CABURET, Institut Jacques Monod, Paris, France
Collaborations antérieures :
- Frédérique DESHAYES, Institut Jacques Monod, Paris, France
- Farida DJOUAD, IRMB, Montpellier, France
- Shoumo BHATTACHARYA, Radcliffe Dept of Medicine, University of Oxford, Oxford, UK
- Renata KOZYRAKI, Centre de Recherche des Cordeliers, Paris, France
- Michel COHEN-TANNOUDJI, Institut Pasteur, Paris, France
- Sophie CREUZET, Institut des Neurosciences Paris-Saclay, Université Paris-Saclay, Saclay, France
- An ZWIJSEN, Dept for Human Genetics, KU Leuven, Leuven, Belgium
- Susana M. CHUVA DE SOUSA LOPES, Leiden University Medical Centre, Leiden, The Netherlands
- Sascha OTT, Warwick Systems Biology Centre, University of Warwick, Coventry, UK
- Kirstie A. LAWSON, MRC Human Genetics Unit, University of Edinburgh, Edinburgh, UK
- Georgy KOENTGES, Wolfson Institute for Biomedical Research, University College London, London, UK
- Arnaud DUBOIS, Laboratoire d’Optique Physique, ESPCI, Paris, France
- Sylvie MAZAN, Développement et Évolution des Vertébrés, Université Paris-Sud, Orsay, France
- Bénédicte DURAND, CGMC, Université Claude Bernard Lyon-1, Villeurbanne
- Christian CIBERT, Institut Jacques Monod, Paris, France
Financements en cours :

Financements antérieurs :



