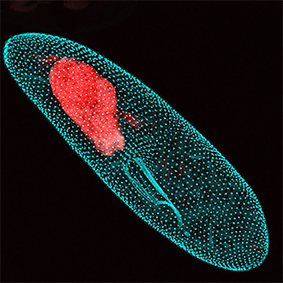
Élimination programmée du génome
Sandra DUHARCOURT
Nos travaux visent à comprendre les mécanismes fondamentaux gouvernant la structure des chromosomes et la stabilité génétique chez les eucaryotes. Pour élucider les mécanismes moléculaires affectant l’intégrité du génome, nous étudions un processus remarquable d’édition du génome programmée au cours du développement chez l’eucaryote unicellulaire Paramecium.
Mots-clés : épigénétique, transposons, ARN non codant, chromatine, évolution du génome, Paramecium
+33 (0)1 57 27 80 09 Contact @sandraduharcourt.bsky.social
Chez Paramecium, au moins un tiers du génome germinal est éliminé au cours du développement. Ce processus implique l’élimination massive et reproductible de séquences répétées d’ADN, telles que les éléments transposables et de 45 000 courtes séquences non codantes, dispersées dans le génome, reliques d‘éléments transposables. Aucun motif de séquence conservé qui pourrait servir de signal de reconnaissance spécifique n’a été identifié parmi les séquences éliminées. Comprendre comment des séquences aussi diverses sont reconnues et excisées reste un défi.
Nos travaux de recherche en cours visent à i) identifier les séquences éliminées et décrire leur trajectoire évolutive, ii) élucider les mécanismes moléculaires de l’élimination programmée d’ADN et iii) comprendre les possibles fonctions associées à cette réorganisation massive du génome.
Le processus d’élimination implique la formation d’hétérochromatine dirigée par des petits ARN, l’excision des séquences associées à l’hétérochromatine, et la réparation de l’ADN. Les petits ARNs sont produits à partir du génome de la lignée germinale pendant la méiose et guident le dépôt des modifications épigénétiques post-traductionnelles de l’histone H3 (H3K9me3 et H3K27me3) sur les séquences à éliminer dans le noyau somatique en développement, et recrutent specifiquement les machineries de clivage et de réparation de l’ADN.
Nous avons démontré que l’histone méthyltransférase Ezl1 de Paramecium, homologue de la sous-unité catalytique de la PRC2 des mammifères, catalyse H3K27me3 et H3K9me3 sur les éléments transposables. Nous étudions actuellement comment le complexe associé à Ezl1 est recruté sur ces éléments, et comment les modifications post-traductionnelles de l’histone H3 déclenchent le clivage et la réparation de l’ADN. Nous combinons un large panel d’approches moléculaires, cellulaires, de séquençage à haut débit, génétiques et biochimiques pour étudier le rôle des facteurs chromatiniens dans le contrôle épigénétique de l’élimination de l’ADN.
En utilisant une approche innovante de cytométrie de flux pour purifier les noyaux, nous avons séquencé les génomes de neuf espèces de Paramecium pour la génomique comparative. Ces ressources nous permettront d’élucider la structure des chromosomes de Paramecium, les trajectoires évolutives des séquences éliminées et leurs mécanismes de reconnaissance et d’élimination.
Membres
 Louise ABBOU, Ingénieur en biologie, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B
Louise ABBOU, Ingénieur en biologie, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B Gaelle ALLEON, Stagiaire, DUHARCOURT LAB
Gaelle ALLEON, Stagiaire, DUHARCOURT LAB Thomas BALAN, Doctorant, DUHARCOURT LAB+33 (0)1 57 27 80 00, bureau 554B
Thomas BALAN, Doctorant, DUHARCOURT LAB+33 (0)1 57 27 80 00, bureau 554B Olivia CHARMANT, Postdoctorante, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B
Olivia CHARMANT, Postdoctorante, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B Marina GIOVANNETTI, Doctorante, DUHARCOURT LAB+33 (0)157278009, bureau 554B
Marina GIOVANNETTI, Doctorante, DUHARCOURT LAB+33 (0)157278009, bureau 554B Leticia KOCH LERNER, Enseignant-chercheur, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B
Leticia KOCH LERNER, Enseignant-chercheur, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B Julien RICHARD ALBERT, Postdoctorant, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B
Julien RICHARD ALBERT, Postdoctorant, DUHARCOURT LAB+33 (0)1 57 27 80 09, bureau 554B Razika ZENAKHRI, Stagiaire, DUHARCOURT LAB
Razika ZENAKHRI, Stagiaire, DUHARCOURT LAB
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
- Olivia Charmant, Julita Gruchota, Olivier Arnaiz, Katarzyna P Nowak, Nicolas Moisa
 n, Coralie Zangarelli, Mireille Bétermier, Anna Anielska-Mazur, Véronique Legros, Guillaume Chevreux, Jacek K Nowak, Sandra Duharcourt, The PIWI-interacting protein Gtsf1 controls the selective degradation of small RNAs in Paramecium, Nucleic Acids Research, 2024; https://doi.org/10.1093/nar/gkae1055
n, Coralie Zangarelli, Mireille Bétermier, Anna Anielska-Mazur, Véronique Legros, Guillaume Chevreux, Jacek K Nowak, Sandra Duharcourt, The PIWI-interacting protein Gtsf1 controls the selective degradation of small RNAs in Paramecium, Nucleic Acids Research, 2024; https://doi.org/10.1093/nar/gkae1055
- Miro-Pina, C., Arnaiz, O., Vanssay, A. de, Frapporti, A., Charmant, O., Humbert, A., Lhuillier-Akakpo, M., & Duharcourt, S. (2023). Non-catalytic function of PRC2 in the control of small RNA dynamics during programmed genome elimination in Paramecium. bioRxiv. https://doi.org/10.1101/2023.07.04.547679 (preprint)
- Miró-Pina C, Charmant O, Kawaguchi T, Holoch D, Michaud A, Cohen I, Humbert A, Jaszczyszyn Y, Chevreux G, Del Maestro L, Ait-Si-Ali S, Arnaiz O, Margueron R, Duharcourt S. (2022) Paramecium Polycomb Repressive Complex 2 physically interacts with the small RNA binding PIWI protein to repress transposable elements.
 Dev Cell. 2022 Apr 6;S1534-5807(22)00208-8. doi: 10.1016/j.devcel.2022.03.014. PMID: 35429435
Dev Cell. 2022 Apr 6;S1534-5807(22)00208-8. doi: 10.1016/j.devcel.2022.03.014. PMID: 35429435 - Gnan S#, Matelot M#, Weiman M, Arnaiz O, Guérin F, Sperling L, Bétermier M, Thermes C, Chen CL*, Duharcourt S*. (2022) GC content but not nucleosome positioning directly contributes to intron-splicing efficiency in Paramecium. Genome Res. 2022 Mar 9;gr.276125.121. doi: 10.1101/gr.276125.121. PMID: 35264448
- Déléris A, Berger F, Duharcourt S*. (2021). Role of Polycomb in the control of transposable elements. Trends Genet. 2021 Oct;37(10):882-889. doi: 10.1016/j.tig.2021.06.003. PMID: 34210514 Review.
- Sellis S#, Guérin F#, Arnaiz O, Pett W, Lerat E, Boggetto N, Krenek S, Berendonk T, Couloux A, Aury J-M, Labadie K, Malinsky S, Bhullar S, Meyer E, Sperling L, Duret L*, Duharcourt S*. (2021). Massive colonization of protein-coding exons by selfish genetic elements in Paramecium germline genomes. PLoS Biol 19(7): e3001309. https://doi.org/10.1371/journal.pbio.3001309 PMID: 34324490
- https://insb.cnrs.fr/fr/cnrsinfo/invasion-massive-delements-genetiques-egoistes-dans-les-genes-de-paramecie
- Primer: Feng Y, Landweber LF (2021) Transposon debris in ciliate genomes. PLoS Biol 19(8): e3001354. https://doi.org/10.1371/journal.pbio.3001354
- Vanssay A#, Touzeau A#, Arnaiz O, Frapporti A, Phipps J, Duharcourt S. (2020). The Paramecium histone chaperone Spt16-1 is required for Pgm endonuclease function in programmed genome rearrangements. PLoS Genet. 2020 Jul 23;16(7):e1008949. doi: 10.1371/journal.pgen.1008949. PMID: 32702045
- Frapporti A#, Miró-Pina C#, Arnaiz O, Holoch D, Kawaguchi T, Humbert A, Eleftheriou E, Lombard B, Loew D, Sperling L, Guitot K, Margueron R, Duharcourt S. (2019). The Polycomb protein Ezl1 mediates H3K9 and H3K27 methylation to repress transposable elements in Paramecium. Nat Commun. 2019 Jun 20;10(1):2710. doi: 10.1038/s41467-019-10648-5. PMID: 31221974
- Guérin F, Arnaiz O, Boggetto N, Denby Wilkes C, Meyer E, Sperling L and Duharcourt S. (2017). Flow cytometry sorting of nuclei enables the first global characterization of Paramecium germline DNA and transposable elements. BMC Genomics. 2017 Apr 26;18(1):327. PMID: 28446146
- Lhuillier-Akakpo M, Guérin F, Frapporti A and Duharcourt S. (2016). DNA deletion as a mechanism for developmentally programmed centromere loss. Nucleic Acids Research. 2016 Feb 29;44(4):1553-65. doi: 10.1093/nar/gkv1110. PMID: 26503246
- Lhuillier-Akakpo M#, Frapporti A#, Denby Wilkes C, Matelot M, Vervoort M, Sperling L, Duharcourt S. (2014) Local effect of enhancer of zeste-like reveals cooperation of epigenetic and cis-acting determinants for zygotic genome rearrangements. PLoS Genet. 2014 Sep 25;10(9):e1004665. doi: 10.1371/journal.pgen.1004665. PMID: 25254958
- Singh DP, Saudemont B, Guglielmi G, Arnaiz O, Goût JF, Prajer M, Potekhin A, Przybòs E, Aubusson-Fleury A, Bhullar S, Bouhouche K, Lhuillier-Akakpo M, Tanty V, Blugeon C, Alberti A, Labadie K, Aury JM, Sperling L, Duharcourt S, Meyer E. (2014). Genome-defence small RNAs exapted for epigenetic mating-type inheritance. 2014 May 22;509(7501):447-52. doi: 10.1038/nature13318. PMID: 24805235
Publication
Preprint
Revue
Chapitre de livre
- Marina Giovannetti 2024 – financement Université Paris Cité (ED BioSPC)
- Thomas Balan 2021 – financement Université Paris Cité (ED BioSPC) et FRM
- Olivia Charmant 2019 – 2023 – financements Université Paris Cité et FRM
- Caridad Miro-Pina 2016-2020- financements Université Paris Cité et Fondation ARC
- Amandine Touzeau 2014-2018 – financements Université Paris Cité et Fondation ARC
- Andrea Frapporti 2012-2016 – financements Université Paris Cité et FRM
- Maoussi Lhuillier-Akakpo 2010-2014 – financements Sorbonne Université et FRM
- Raphaël Margueron (Institut Curie, Paris, France)
- Olivier Arnaiz (I2BC, Gif, France)
- Slimane Ait-Si-Ali (EDC, Universite Paris Cité, France)
- Eric Meyer (IBENS, Paris, France)
- Laurent Duret (LBBE, Lyon, France)
- Mireille Bétermier (I2BC, Gif, France)
- Chunlong Chen (Institut Curie, Paris, France)
- Hadi Quesneville (INRA, Versailles, France)
- Karine Labadie/ Jean-Marc Aury (Génoscope, Evry, France)
- Thomas Berendonk (TU, Dresde, Allemagne)
- Ewa Przybòs/Natalia Sawka (ISEA PAS, Cracovie, Pologne)
- Alexey Potekhin (SPBU, St. Petersbourg, Russie)
- Matthieu Defrance (ULB, Bruxelles, Belgique)
- Julita Gruchota, Jacek Nowak (IBB, Varsovie, Pologne)

ANR LaMarque 2018-2022
ANR POLYCHROME 2019-2023
ANR SELECTION 2024-2026
 LabEx Who Am I? 2021-2023
LabEx Who Am I? 2021-2023

Equipe labellisée FRM 2022-2025

04/07/2023 – Sandra Duharcourt, élection membre EMBO
04/04/2022 – Sandra Duharcourt, Médaille d’argent du CNRS , Vidéo sur la chaîne Youtube CNRS