Division cellulaire et reproduction
Julien DUMONT
 Pendant la division cellulaire, les chromosomes, supports de l’information génétique, sont répartis en deux lots égaux entre les cellules filles. Au cours du développement, l’aneuploïdie, qui correspond à la présence d’un nombre incorrect de chromosomes, conduit à la formation d’embryons généralement non viables ou présentant des défauts sévères de développement. Notre projet vise à étudier les mécanismes garantissant la formation d’ovocytes et d’embryons contenant un nombre correct de chromosomes. Nous étudions pour cela les mécanismes d’assemblage et le fonctionnement de la machinerie cellulaire permettant la répartition correcte des chromosomes en méiose et en mitose. Notre approche est multidisciplinaire et s’appuie sur des outils génétiques, biochimiques, cellulaires et microscopiques de pointe.
Pendant la division cellulaire, les chromosomes, supports de l’information génétique, sont répartis en deux lots égaux entre les cellules filles. Au cours du développement, l’aneuploïdie, qui correspond à la présence d’un nombre incorrect de chromosomes, conduit à la formation d’embryons généralement non viables ou présentant des défauts sévères de développement. Notre projet vise à étudier les mécanismes garantissant la formation d’ovocytes et d’embryons contenant un nombre correct de chromosomes. Nous étudions pour cela les mécanismes d’assemblage et le fonctionnement de la machinerie cellulaire permettant la répartition correcte des chromosomes en méiose et en mitose. Notre approche est multidisciplinaire et s’appuie sur des outils génétiques, biochimiques, cellulaires et microscopiques de pointe.
Mots-clés : Division cellulaire, Méiose/Mitose, Cytosquelette, Ségrégation des chromosomes, Développement embryonnaire.
+33 (0)1 57 27 80 49 Contact
Environ 15% des couples sont confrontés à un problème d’infertilité. Les causes potentielles en sont la mauvaise qualité des cellules sexuelles ou gamètes (spermatozoïde et ovocyte), ou des défauts lors du développement de l’embryon. Un enjeu majeur de la biologie de la reproduction est d’en comprendre les mécanismes.
Au cours de la division cellulaire, les chromosomes, supports de l’information génétique, sont répartis en deux lots égaux entre les cellules filles. La répartition fidèle des chromosomes est un élément fondamental de la stabilité génétique des cellules et des organismes. L’aneuploïdie, qui correspond à la présence d’un nombre incorrect de chromosomes, conduit à la formation d’embryons généralement non viables ou présentant des défauts sévères de développement. Notre projet vise à étudier les mécanismes garantissant la formation d’ovocytes et d’embryons contenant un nombre correct de chromosomes, élément essentiel à la reproduction.
Nous étudions pour cela les mécanismes d’assemblage et le fonctionnement de la machinerie cellulaire permettant la répartition correcte des chromosomes en méiose et en mitose. Cette machinerie se compose d’un fuseau de division formé de microtubules (en vert sur la figure 1) dont certains contactent les chromosomes (magenta). Les interactions entre microtubules du fuseau et chromosomes sont au centre du processus de ségrégation des chromosomes et sont régulées de façon différente en méiose et en mitose.
Notre approche est multidisciplinaire et s’appuie sur des outils génétiques, biochimiques, cellulaires et microscopiques de pointe. Notre modèle d’étude favori est le ver nématode Caenorhabditis elegans qui partage avec les mammifères la grande majorité des gènes et des mécanismes clefs de la reproduction et du développement. Nous étudions également des vers nématodes « non modèles » afin de développer une compréhension évolutive des mécanismes de la division cellulaire dans le contexte de la reproduction.
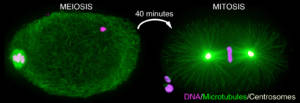
Figure 1 : Marquages immunofluorescents d’un fuseau méiotique d’ovocyte (à gauche) et d’un fuseau mitotique de zygote (à droite) chez C. elegans. Les microtubules sont en vert, les chromosomes en magenta et les deux centrosomes qui forment les pôles du fuseau en mitose sont en jaune. Notez l’absence de centrosomes aux pôles du fuseau méiotique et sa forme caractéristique « en tonneau ».
Axe 1/ Divisions méiotique de l’ovocyte :
En mitose, les microtubules du fuseau sont assemblés à partir des centrosomes (Figure 1, jaune). Dans les ovocytes, les centrosomes sont éliminés précocement. L’assemblage des fuseaux dans les ovocytes met donc en jeu des mécanismes atypiques mal connus. Nous avons mis au point une approche de microscopie in utero sur nématodes vivants qui nous a permis de décrire les étapes clefs de la formation des fuseaux acentrosomaux dans l’ovocyte de C. elegans (Vidéo 1).
Vidéo 1
Afin de déterminer les acteurs impliqués, nous combinons cette méthode d’imagerie confocale avec une approche de perte de fonction systématique des protéines associées aux microtubules et des moteurs microtubulaires. Nous utilisons également une approche in vitro de microscopie TIRF (Total Internal Reflection Microscopy) sur protéines recombinantes et microtubules individuels (Figure 2). Notre but est de déterminer le réseau d’acteurs nécessaire à la formation d’un fuseau en absence de centrosome, ainsi que de mettre en évidence les interactions fonctionnelles entre ces acteurs.

Figure 2
Axe 2/ Ségrégation des chromosomes dans l’ovocyte :
Les fuseaux acentrosomaux ont une forme caractéristique dite « en tonneau » (Figure 1), avec des pôles arrondis, due à l’absence de microtubules astraux (microtubules qui, en mitose, connectent normalement les pôles des fuseaux au cortex cellulaire). Cette forme atypique induit la mise en jeu de mécanismes spécifiques de ségrégation des chromosomes méiotiques. Par une approche de microscopie multimodale, combinant microscopie optique live d’ovocytes de C. elegans, ablation laser sur ovocytes vivants et microscopie électronique en tomographie, nous avons mis en évidence un mécanisme original de ségrégation des chromosomes « par poussée » exercée par les microtubules (Vidéo 2 et Figure 3). Nous combinons les différentes approches de microscopie avec des pertes de fonction ciblées d’acteurs clefs afin d’en déterminer les mécanismes moléculaires.
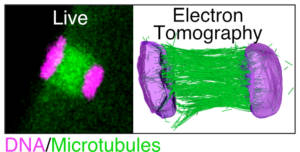
Figure 3
Nous avons, par ailleurs, montré que la ségrégation « par poussée » ne dépend pas strictement des kinétochores, complexes multiprotéiques s’assemblant sur les chromosomes pour permettre la liaison des microtubules. Néanmoins ces derniers sont importants pour assurer la fidélité du mécanisme de ségrégation et éviter l’aneuploïdie ovocytaire. Nous avons récemment mis au point une méthode de microscopie live de deux canaux de fluorescence simultanément avec scan rapide en Z (Figure 4).
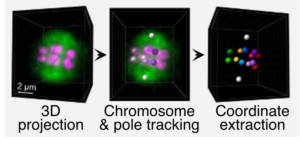
Figure 4
Cette méthode permet d’acquérir la totalité du volume de fuseau méiotique et des chromosomes en cours de ségrégation pendant toute la première division méiotique. Nous pouvons ainsi suivre la position de chaque chromosome individuellement pendant les étapes d’orientation, d’alignement et de ségrégation sur le fuseau méiotique. Cette approche nous permet donc de réaliser une analyse quantitative de la fonction de protéines candidates et d’en déterminer l’importance pour la ségrégation des chromosomes dans l’ovocyte de C. elegans.
Axe 3/ Évolution des mécanismes de la division méiotique chez les nématodes
Des défauts de ségrégation des chromosomes au cours de la méiose peuvent entraîner l’aneuploïdie des gamètes et de l’embryon, qui est une cause majeure de malformations congénitales et d’avortements spontanés chez l’homme. Cependant, bien qu’ils soient essentiels à la reproduction des organismes multicellulaires et donc à la continuité des espèces, les principes clés qui régissent la méiose, et en particulier la ségrégation chromosomique méiotique dans les ovocytes, sont encore mal compris. Une des principales raisons de ce manque de connaissances est la diversité des scénarios moléculaires qui ont été adoptés au cours de l’évolution pour réguler la ségrégation des chromosomes, en particulier dans les ovocytes. En effet, bien que la méiose soit hautement conservée chez les eucaryotes, les déviations de la “norme” sont omniprésentes et peuvent fournir des informations importantes sur la signification évolutive des mécanismes de la méiose. Il est donc de la plus haute importance d’apprécier la véritable diversité des caractéristiques méiotiques dans la nature, en considérant à la fois des organismes modèles bien caractérisés (comme C. elegans) et des organismes non modèles. Les nématodes représentent une situation idéale à cet égard : leur remarquable adaptabilité à diverses niches écologiques s’est accompagnée d’une extrême plasticité dans l’organisation du génome mais aussi dans les stratégies reproductives et méiotiques. Nous avons récemment commencé l’analyse de la méiose ovocytaire dans des espèces de nématodes non modèles présentant des caractéristiques spécifiques et atypiques susceptibles d’avoir un impact sur la ségrégation des chromosomes méiotiques (Vidéo 3).
Axe 4/ La première division mitotique du zygote de c.elegans
Dans le zygote de C. elegans, en mitose, les microtubules émanant des pôles du fuseau contactent soit le cortex embryonnaire (microtubules astraux), soit les chromosomes via les kinétochores (microtubules kinétochoriens). Les forces de traction exercées par les microtubules astraux sont transmises aux chromosomes par les microtubules kinétochoriens permettant ainsi la ségrégation des chromatides sœurs en anaphase (Vidéo 4).
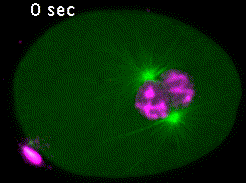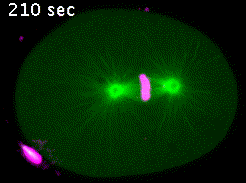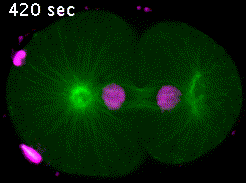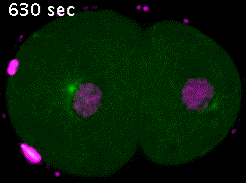
Les kinétochores sont des complexes multiprotéiques constitués de plusieurs dizaines de protéines différentes. Parmi celles-ci, la kinase BUB-1, les protéines HCP-½ et la protéine de liaison aux microtubules CLS-2, forment un module impliqué dans le contrôle de la dynamique des microtubules kinétochoriens. Nous avons mis en évidence les rôles de ce module dans l’alignement et l’attachement correctes des chromosomes en métaphase, et dans la formation du fuseau central de microtubule en anaphase. Nous poursuivons l’analyse des différentes fonctions du module BUB-1/HCP-½/CLS-2 au cours de la première division mitotique du zygote de C. elegans. Nous développons, pour cela, une approche de mutagenèse ciblée couplée à la microscopie confocale sur embryons vivants. Cette approche nous permet de réaliser une analyse quantitative du rôle des différents acteurs à travers les étapes de la mitose (Figure 5).
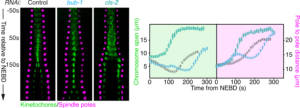
Figure 5
Membres
 Laura GUEDON, Ingénieur technicien, DUMONT LAB
Laura GUEDON, Ingénieur technicien, DUMONT LAB Leo LENHARDT, Stagiaire, DUMONT LAB
Leo LENHARDT, Stagiaire, DUMONT LAB Gilliane MATON, Enseignant-chercheur, DUMONT LAB+33 (0)1 57 27 80 47, bureau 402B
Gilliane MATON, Enseignant-chercheur, DUMONT LAB+33 (0)1 57 27 80 47, bureau 402B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr

El Mossadeq L, Bellutti L, Le Borgne R, Canman JC, Pintard L, Verbavatz J-M, Askjaer P & Dumont J. An interkinetic envelope surrounds chromosomes between meiosis I and II in C. elegans oocytes. J Cell Biol (accepted, 2024)
Belluti L, Macaisne N, El Mossadeq L, Ganeswaran T, Canman JC & Dumont J. Regulation of outer kinetochore assembly during meiosis I and II by CENP-A and KNL-2/M18BP1 in C. elegans oocytes. Current Biology S0960-9822(24)01215-6 doi: 10.1016/j.cub.2024.09.004 (2024)
Perrier A, Guiglielmoni N, Naquin D, Gorrichon K, Thermes C, Lameiras S, Dammermann A, Schiffer PH, Brunstein M, Canman JC & Dumont J. Maternal inheritance of functional centrioles in two parthenogenic nematodes. Nat Comm 15(1):6042. doi: 10.1038/s41467-024-50427-5 (2024)
Gareil N*, Gervais A*, Macaisne N, Chevreux G, Canman JC, Andreani J & Dumont J. An unconventional TOG domain is required for CLASP localization. Current Biology, 33(16):3522-3528.e7 doi: 10.1016/j.cub.2023.07.009 (2023)
Pitayu-Nugroho L*, Aubry M*, Laband K, Geoffroy H, Ganeswaran T, Primadhanty A, Canmam JC & Dumont J. Kinetochore component function in C. elegans oocytes revealed by 4D tracking of holocentric chromosomes. Nat Comm, 14, 4032. doi: 10.1038/s41467-023-39702-z (2023)
Macaisne N*, Bellutti L*, Laband K*, Edwards F*, Pitayu-Nugroho L, Gervais A, Ganeswaran T, Geoffroy H, Maton G, Canman JC, Lacroix B & Dumont J. Synergistic stabilization of microtubules by BUB-1, HCP-1 and CLS-2 controls microtubule pausing and meiotic spindle assembly. eLife, 12:e82579. doi: 10.7554/eLife.82579 (2023)
Edwards F, Maton G, Gareil N, Canman JC & Dumont J. BUB-1 promotes amphitelic chromosome biorientation via multiple activities at the kinetochore. eLife 2018;7:e40690 doi: 10.7554/eLife.40690 (2018)
Lacroix B, Letort G, Pitayu L, Sallé J, Stefanutti M, Maton G, Ladouceur AM, Canman JC, Maddox PS, Maddox AS, Minc N, Nédélec F & Dumont J. Microtubule dynamics scale with cell size to set spindle length and assembly timing. Dev. Cell, 45, (4):496-511 doi: 10.1016/j.devcel.2018.04.022 (2018) Highlighted in Dev Cell 45:421-423, (2018) F1000: https://f1000-com.insb.bib.cnrs.fr/prime/733268901#eval793546852
Laband K, Le Borgne R, Edwards F, Stefanutti M, Canman JC, Verbavatz J-M & Dumont J. Chromosome segregation occurs by microtubule pushing in oocytes. Nat Commun, 14;8(1):1499 doi: 10.1038/s41467-017-01539-8 (2017)
Gigant E*, Stefanutti M*, Laband K, Gluszek-Kustusz A, Edwards F, Maton G, Lacroix B, Canman JC, Welburn J & Dumont J. Inhibition of ectopic microtubule assembly by the kinesin-13 KLP-7MCAK prevents chromosome segregation and cytokinesis defects in oocytes. Development, 144(9):1674-1686, doi: 10.1242/dev.147504 (2017)
Maton G*, Edwards F*, Lacroix B, Stefanutti M, Laband K, Lieury T, Kim T, Espeut J, Canman JC, & Dumont J. Kinetochore components are required for central spindle assembly. Nat Cell Biol, 17(5):697-705 doi: 10.1038/ncb3150 (2015) Dispatched in Current Biology 25, R554–R557, (2015)
Dumont J, Oegema K & Desai A. A kinetochore-independent mechanism drives anaphase chromosome separation during acentrosomal meiosis. Nat Cell Biol,12(9):894-901 doi: 10.1038/ncb2093 (2010)
Publications
2024
El Mossadeq L, Bellutti L, Le Borgne R, Canman JC, Pintard L, Verbavatz J-M, Askjaer P & Dumont J. An interkinetic envelope surrounds chromosomes between meiosis I and II in C. elegans oocytes. J Cell Biol (accepted, 2024)
Xie J, Najafi J, Nommick A, Lederer L, Salle J, Dmitrieff S, Lacroix B, Dumont J. & Minc N. Cell shape modulates mitotic spindle positioning forces via intracellular hydrodynamics. Current Biology (accepted, 2024)
Belluti L, Macaisne N, El Mossadeq L, Ganeswaran T, Canman JC & Dumont J. Regulation of outer kinetochore assembly during meiosis I and II by CENP-A and KNL-2/M18BP1 in C. elegans oocytes. Current Biology S0960-9822(24)01215-6 doi: 10.1016/j.cub.2024.09.004 (2024)
Perrier A, Guiglielmoni N, Naquin D, Gorrichon K, Thermes C, Lameiras S, Dammermann A, Schiffer PH, Brunstein M, Canman JC & Dumont J. Maternal inheritance of functional centrioles in two parthenogenic nematodes. Nat Comm 15(1):6042. doi: 10.1038/s41467-024-50427-5 (2024)
Connors CQ, Martin SL, Dumont J, Shirasu-Hiza M, Canman JC. Cell type-specific regulation by different cytokinetic pathways in the early embryo. MicroPubl Biol. Oct 22;2024:10.17912/micropub.biology.001316. doi:10.17912/micropub.biology.001316 (2024)
Connors CQ, Mauro MS, Tristian Wiles J, Countryman AD, Martin SL, Lacroix B, Shirasu-Hiza M, Dumont J, Kasza KE, Davies TR & Canman JC. Germ fate determinants protect germ precursor cell division by restricting septin and anillin levels at the division plane. MBoC 35(7), doi: 10.1101/2023.11.17.566773 (2024)
2023
Gareil N*, Gervais A*, Macaisne N, Chevreux G, Canman JC, Andreani J & Dumont J. An unconventional TOG domain is required for CLASP localization. Current Biology, 33(16):3522-3528.e7 doi: 10.1016/j.cub.2023.07.009 (2023)
Pitayu-Nugroho L*, Aubry M*, Laband K, Geoffroy H, Ganeswaran T, Primadhanty A, Canmam JC & Dumont J. Kinetochore component function in C. elegans oocytes revealed by 4D tracking of holocentric chromosomes. Nat Comm, 14, 4032. doi: 10.1038/s41467-023-39702-z (2023)
Macaisne N*, Bellutti L*, Laband K*, Edwards F*, Pitayu-Nugroho L, Gervais A, Ganeswaran T, Geoffroy H, Maton G, Canman JC, Lacroix B & Dumont J. Synergistic stabilization of microtubules by BUB-1, HCP-1 and CLS-2 controls microtubule pausing and meiotic spindle assembly. eLife, 12:e82579. doi: 10.7554/eLife.82579 (2023)
Lignieres L, Senecaut N, Dang T, Bellutti L, Hamon M, Terrier S, Legros V, Chevreux G, Lelandais G, Mege RM, Dumont J & Camadro JM. Extending the range of SLIM-labeling applications: From human cell lines in culture to Caenorhabditis elegans whole-organism labeling. J Proteome Research, 22(3):996-1002. doi: 10.1021/acs.jproteome.2c00699 (2023)
Rocha H, Simões PA, Budrewicz J, Lara-Gonzalez P, Carvalho AX, Dumont J, Desai A, Gassmann R. Nuclear-enriched protein phosphatase 4 ensures outer kinetochore assembly prior to nuclear dissolution. J Cell Biol, 222(3):e202208154 doi: 10.1083/jcb.202208154 (2023)
2022
Hirsch S, Edwards F, Shirasu-Hiza M, Dumont J & Canman JC. Functional midbody assembly in the absence of a central spindle. J Cell Biol, 221(3):e202011085 doi: 10.1083/jcb.202011085 (2022)
2019
Cabral G, Laos T, Dumont J, & Dammermann A. Differential requirements for centrioles in mitotic centrosome growth and maintenance. Dev. Cell, 50, (3):355-366 doi: 10.1016/j.devcel.2019.06.004 (2019)
2018
Edwards F, Maton G, Gareil N, Canman JC & Dumont J. BUB-1 promotes amphitelic chromosome biorientation via multiple activities at the kinetochore. eLife 2018;7:e40690 doi: 10.7554/eLife.40690 (2018)
Hirsch SM, Sundaramoorthy S, Davies T, Zhuravlev Y, Waters JC, Shirasu-Hiza M, Dumont J & Canman JC. FLIRT: Fast Local InfraRed Thermogenetics for subcellular control of protein function. Nature Methods 2018 Nov;15(11):921-923 doi: 10.1038/s41592-018-0168-y (2018)
Davies T, Kim HX, Romano Spica N, Lesea-Pringle BJ, Dumont J, Shirasu-Hiza M & Canman JC. Cell-intrinsic and extrinsic mechanisms promote cell-type specific cytokinetic diversity. eLife 2018;7:e36204 doi: 10.7554/eLife.36204 (2018)
Lacroix B, Letort G, Pitayu L, Sallé J, Stefanutti M, Maton G, Ladouceur AM, Canman JC, Maddox PS, Maddox AS, Minc N, Nédélec F & Dumont J. Microtubule dynamics scale with cell size to set spindle length and assembly timing. Dev. Cell, 45, (4):496-511 doi: 10.1016/j.devcel.2018.04.022 (2018) Highlighted in Dev Cell 45:421-423, (2018) F1000: https://f1000-com.insb.bib.cnrs.fr/prime/733268901#eval793546852
Berto A, Yu J, Morchoisne-Bolhy S, Bertipaglia C, Vallee R, Dumont J, Ochsenbein F, Guerois R & Doye V. Disentangling the molecular determinants for Cenp-F localization to nuclear pores and kinetochores. EMBO Report 19(5). doi: 10.15252/embr.201744742 (2018)
2017
Laband K, Le Borgne R, Edwards F, Stefanutti M, Canman JC, Verbavatz J-M & Dumont J. Chromosome segregation occurs by microtubule pushing in oocytes. Nat Commun, 14;8(1):1499 doi: 10.1038/s41467-017-01539-8 (2017)
Luscan R, Mechaussier S, Paul A, Tian G, Gérard X, Loundon N, Defoort-Delhemmes S, Audo I, Dumont J, Goudin N, Garfa-Traoré M, Bras M, Pouliet A, Attié-Bittach T, Boddaert N, Lyonnet S, Kaplan J, Cowan NJ, Rozet J-M, Marlin S & Perrault I. Mutations in TUBB4B cause a distinctive sensorineural disease. Am J Hum Genet, 101(6):1006-1012 doi: 10.1016/j.ajhg.2017.10.010 (2017)
Martino L, Morchoisne-Bolhy S, Cheerambathur D, Van Hove L, Dumont J, Joly N, Desai A, Doye V & Pintard L. Channel nucleoporins recruit PLK-1 to nuclear pore complexes to direct nuclear envelope breakdown in C. elegans. Dev. Cell, 43, (2):157-171 doi: 10.1016/j.devcel.2017.09.019 (2017)
Gigant E*, Stefanutti M*, Laband K, Gluszek-Kustusz A, Edwards F, Maton G, Lacroix B, Canman JC, Welburn J & Dumont J. Inhibition of ectopic microtubule assembly by the kinesin-13 KLP-7MCAK prevents chromosome segregation and cytokinesis defects in oocytes. Development, 144(9):1674-1686, doi: 10.1242/dev.147504 (2017)
Zhuravlev Y, Hirsch S, Jordan S, Dumont J, Shirasu-Hiza M & Canman JC. CYK-4 regulates Rac, but not Rho, during cytokinesis. MBoC, 28(9):1258-1270 doi: 10.1091/mbc.E17-01-0020 (2017)
Sundaramoorthy S, Garcia BA, Hirsch S, Park JH, Davies T, Dumont J, Shirasu-Hiza M, Kummel A & Canman JC. Low efficiency upconversion nanoparticles for high-resolution coalignment of near-infrared and visible light paths on a light microscope. ACS Appl. Mater. Interfaces, 9 (9), pp 7929-7940 doi: 10.1021/acsami.6b15322 (2017)
Revues
2024
Lacroix B & Dumont J. Spatial and Temporal Scaling of Microtubules and Mitotic Spindles. Cells, 11(2):248 doi: 10.3390/cells11020248 (2022)
Chapitres de livres
2024
Dumont J & Maton G. 4D microscopy and tracking of chromosomes and the spindle in C. elegans early embryos. Methods Mol Biol (2024)
Chenevert J, Robert MLV, Sallé J, Cacchia S, Lorca T, Castro A, McDougall A, Minc N, Castagnetti S, Dumont J & Lacroix B. Measuring Mitotic Spindle and Microtubule Dynamics in Marine Embryos and Non-model Organisms. Methods Mol Biol, 2024:2740:187-210 doi: 10.1007/978-1-0716-3557-5_12 (2024)
Laband K, Lacroix B, Edwards F, Canman JC & Dumont J. Live imaging of C. elegans oocytes and early embryos. Methods Cell Biol, 145:217-236 doi: 10.1016/bs.mcb.2018.03.025 (2018)
Davies T, Sundaramoorthy S, Jordan SN, Shirasu-Hiza M, Dumont J & Canman JC. Using fast-acting temperature sensitive mutants to study cell division in C. elegans. Methods Cell Biol, 137:283-306 doi: 10.1016/bs.mcb.2016.05.004 (2017)
- Kimberley Laband : 2017
- Frances Edwards : 2018
- Layla El Mossadeq : 2022
- Mélanie Aubry : 2023
- Aurélien Perrier : 2023
- Jean-Marc Verbavatz (IJM)
- Lionel Pintard (IJM)
- Julie C. Canman (Columbia University, NYC, USA)
- Marie Delattre, (ENS, France) ; Peter Askjaer, (CABD, Spain)
- Peter Meister, (University of Bern, Switzerland)
- European Research Council
- Mairie De Paris
- Fondation pour la Recherche Médicale
- Agence Nationale de la Recherche

